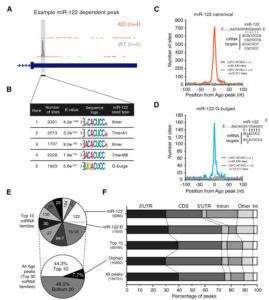
一、RNA-seq分析流程
二、RNA-seq数据质量分析 1.Clean reads数据质量分析 方法描述 :碱基质量值是衡量测序质量的重要指标,碱基质量(Q)与测序错误率(P)密切相关,受测序仪状态,测序试剂质量,样本特性等的影响。
2.GC含量分析
方法描述 :对测序reads中四种碱基的分布比例进行评估,检查是否存在AT、CG分离现象,理论上A与T、C与G的含量在整个测序反应中分别相同,且维持 在稳定水平。
3. 有效长度统计 方法描述 :去掉index序列、建库平衡用随机碱基及截取掉后面低质量的碱基后,我们用获得的clean reads进行有效长度分析。
4. reads冗余度统计 在cDNA文库构建的过程中对捕获的mRNA/ncRNA 进行随机片段化,随后加接头并进行RT-PCR。一个多样性的文库中大多数序列应该只出现一次,低水平的序列冗余度往往表明高水平的靶标序列覆盖度,而高水平的序列冗余度则意味着一定程度上的偏好富集性,如文库构建过程中PCR过度扩增。通常测序深度越高,越容易产生一定程度的重复reads,属于正常的现象。实际操作中,由于数据量较大,为了降低计算中对内存的要求,仅选取了每个文件的前200,000条reads进行分析,认为其可以代表全部序列的冗余度。
PCR duplication level计算方法为 :从测序数据中随机挑选20万reads作为Total Reads,按照如下公式进行计算:PCR duplication level=Duplication Reads/Total Reads
三、RNA-seq结果展示 全基因组定位分析 Reads比对到参考基因组结果 方法描述 :根据不同的基因组的特征,选取相对合适的软件,动植物用HISAT2 (Kim D, Langmead B et al. 2015)、真菌或者基因密度较高的物种用Bowtie2(Langmead and Salzberg 2012),根据需要会设定一定的容错率,将有效测序数据(clean reads)比对到参考基因组上。
Reads在基因组不同区域的分布情况 方法描述 :统计在基因组上有唯一定位的reads在各个区域的分布情况
Reads在染色体上的分布情况 方法描述 :把基因组平均分成100000个bin,根据比在基因组上有唯一定位的(uniquely mapped)reads数,统计落在每个bin中的reads的平均depth,然后取log2,使用circos作图。
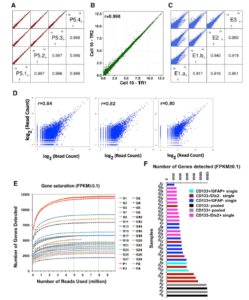
新转录本预测 方法描述 :将所有数据的基因组定位结果合并,用StringTie进行组装,然后用Cuffcompare和已知的基因模型进行比较,发现新的转录区域,并对新转录本的表达水平进行统计分析。图中为已知转录本(annotation)和新转录本(novel)的FPKM累积分布图。
饱和曲线检查 分别对10%,20%,30% … 90%的测序量各自进行基因定量分析,并将以完整测序量分析得到的基因表达水平作为最终表达水平。用各个百分比的数据量得到的基因表达水平和和最终表达水平进行比较,如果差异小于15%,则认为该基因在该数据量条件下被准确定量。
定量饱和曲线检查反映了基因表达水平定量对数据量的要求。表达量高的基因,容易被准确定量,而表达量低的基因,则需要较大的测序量才能被准确定量。
reads在转录起始位点,转录终止位点,起始密码子和终止密码子附近的分布 方法描述 :分别以转录起始位点(TSS)和转录终止位点(TTS)为原点,统计其上下游1kb范围内reads的分布情况,结果如下:
方法描述 :分别以起始密码子(start codon)和终止密码子(stop codon)为原点,统计其上下游1kb范围内reads的分布情况,结果如下:
DEG分析流程
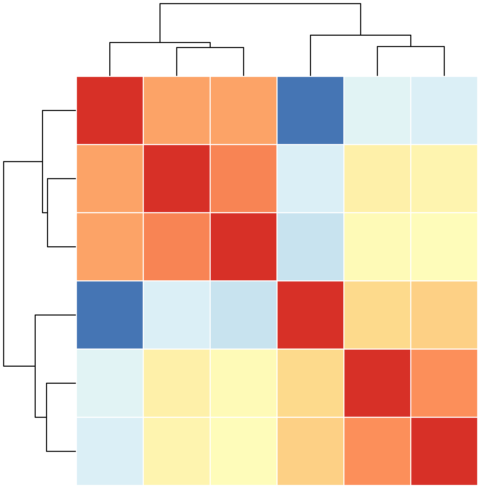
样本相关性分析 方法描述 : 在这部分分析中,我们根据各样本中基因表达情况进行两样本之间的相关性分析,即检查不同样品之间基因表达水平的相关性。如果相关性系数高,说明大部分基因在样本间的表达水平相似,数据均一化程度高;如果相关性系数低,则暗示基因表达模式存在显著差异,或者数据质量有问题。
样本聚类分析 方法描述 :在这部分分析中,我们根据样本相关性系数进行样本间聚类分析。
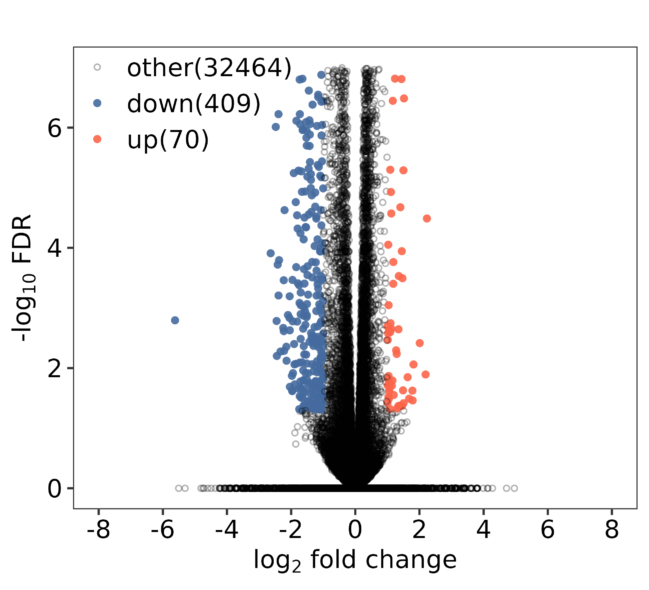
差异基因火山图
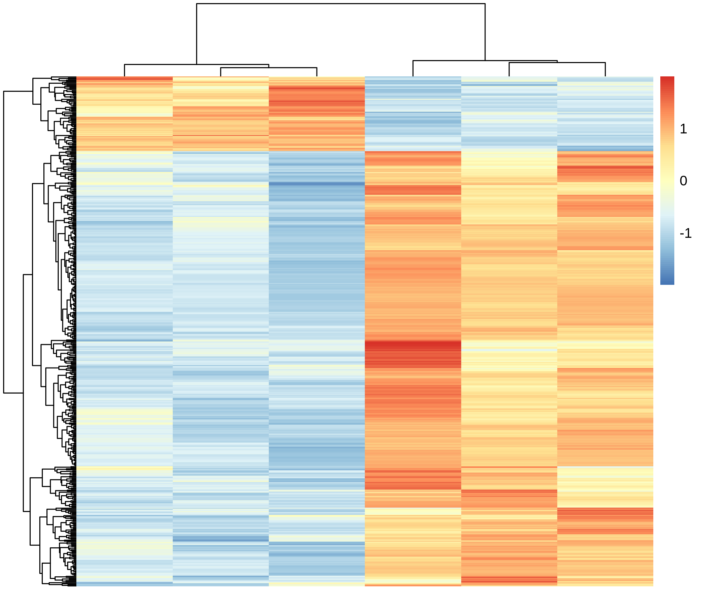
差异基因聚类图 方法描述 :对所有的显著差异表达基因取并集,根据表达量作heatmap。
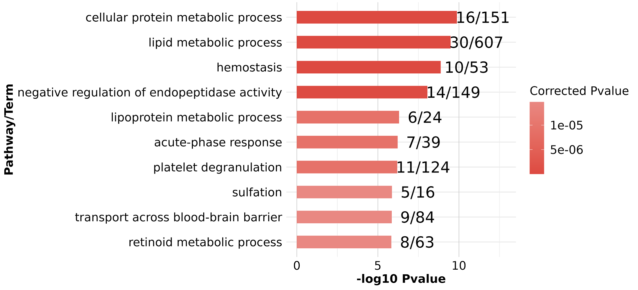
差异基因Gene Ontology 富集分析
差异基因KEGG Pathway富集分析