用别人的数据,发表自己的文章
- 分析模块一
- 分析模块二
- 分析模块三
- 分析模块四
- 分析模块五
- 分析模块六
- 分析模块七
- 分析模块八
- 分析模块九
- 分析模块十
- 单细胞测序大数据分析
- LncRNA+cis-target+WGCNA
- 可变剪接+RBP
- RBP数据包模式+TCGA
- 转录因子+LncRNA/mRNA
- RBP+AS分析(两套GEO数据集分析)
- RBP+LncRNA(白血病、肝癌)
- RBP+免疫浸润分析模块,适用于TCGA的33种癌症
- 铁死亡+免疫浸润分析模块,适用于非TCGA的癌症
- LncRNA/AS+免疫
Bulk-seq大数据亮点挖掘项目成果和案例展示
结果展示(已发表的成果)
分析模块一:使用GEO数据库数据,分析RNA结合蛋白+可变剪接+免疫浸润思路
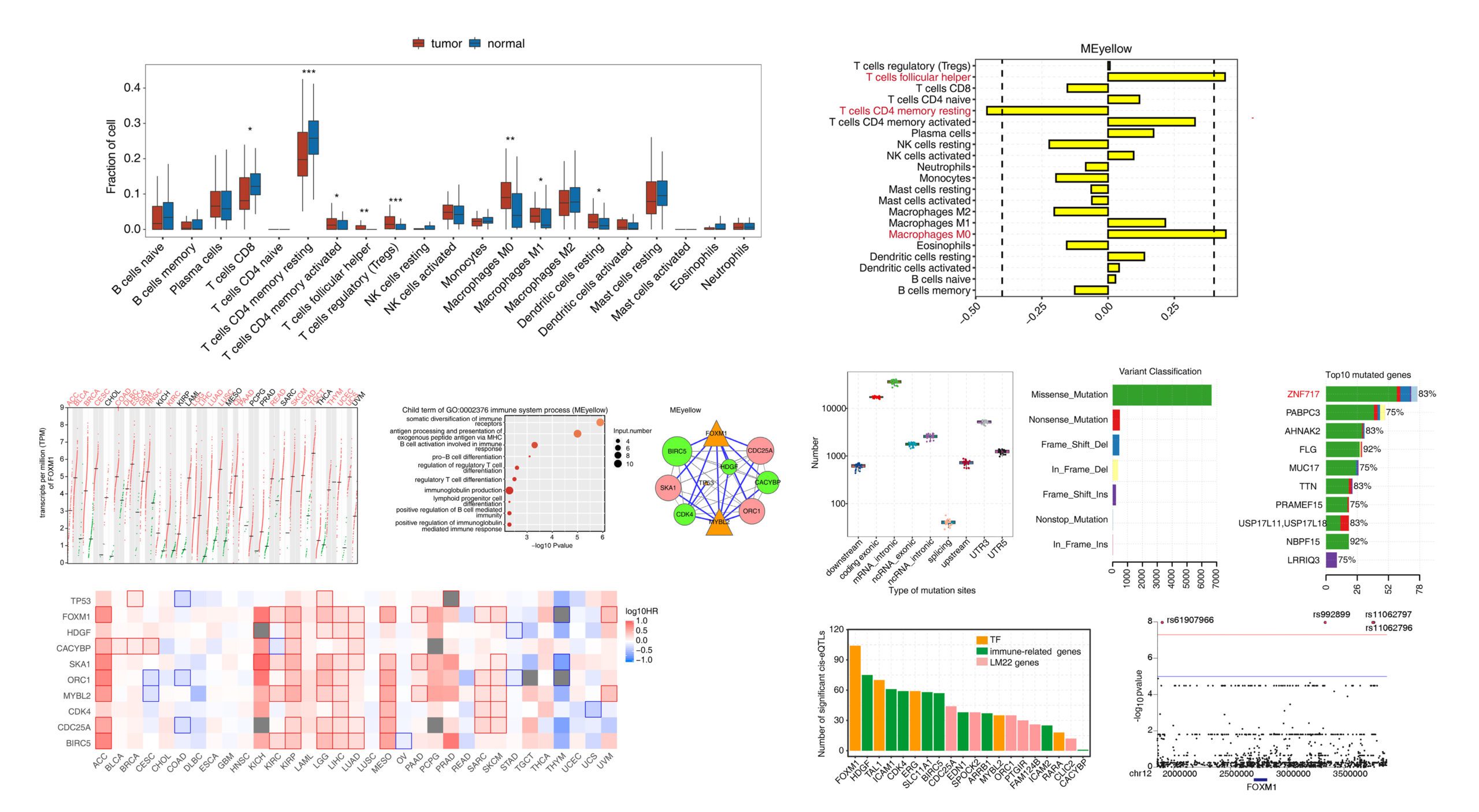
分析模块二:使用TCGA数据库,WGCNA+肿瘤免疫浸润分析
分析模块三:使用自主创新的SUVA分析(剪接位点使用变异分析)数据中可变剪接事件
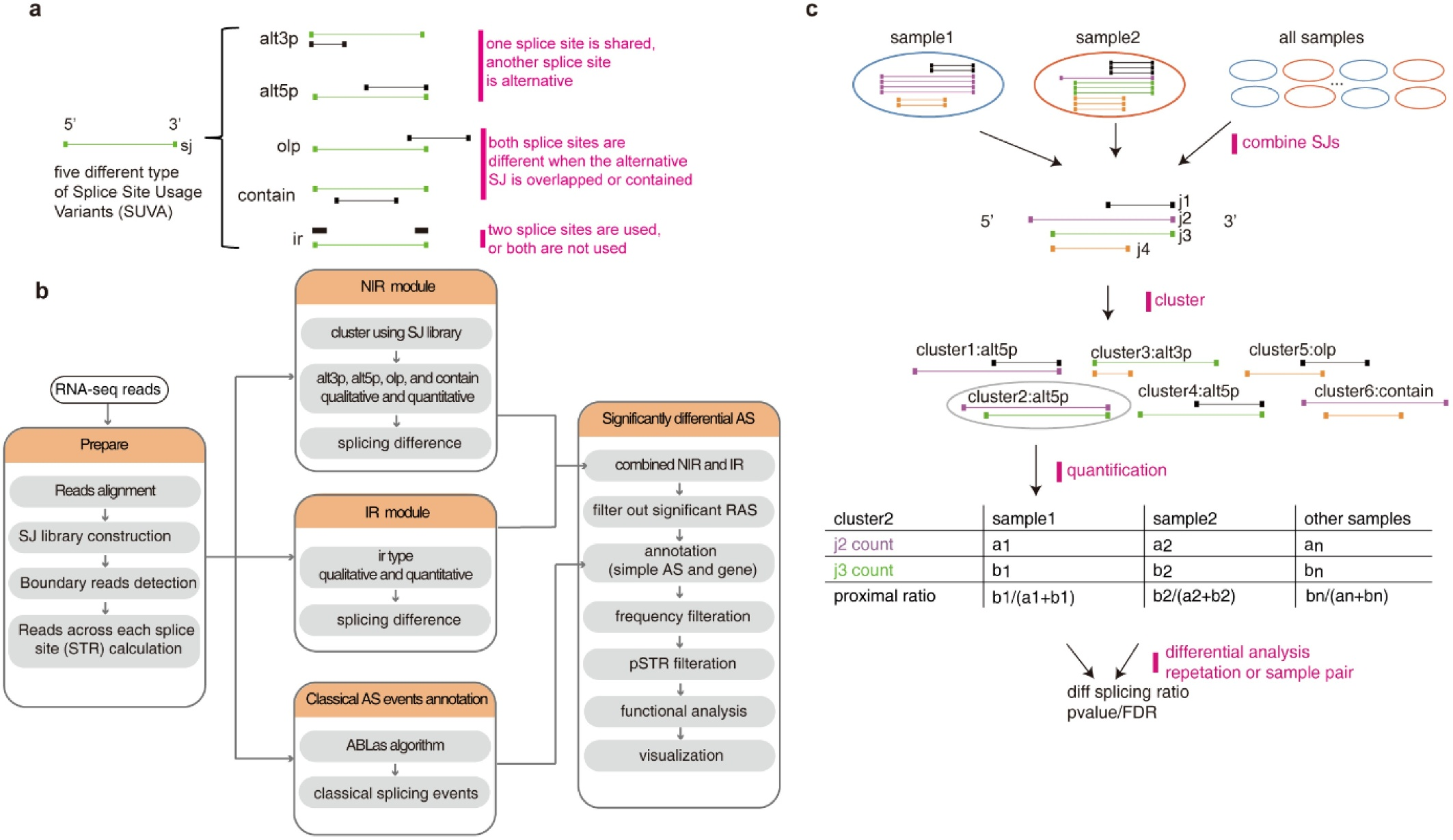
可变剪接(Alternative Splicing, AS)是真核生物中普遍存在的一种关键的转录后过程,通过该过程可以从单个基因中产生多种不同的功能转录本(transcripts)和不同的蛋白质。可变剪接的多样性是构成真核生物体复杂的生物学过程和细胞多样性的重要基础。异常剪接可能导致异常转录和蛋白质产物,这与许多人类疾病密切相关,也是被持续关注的热点领域。因此,更好地描述AS整体特征,更全面地量化特定状态下的这些特征,以及AS变化的特点,对于理解AS在生理和病理状态下的作用至关重要。基于此背景,以及我们在可变剪接调控领域的长年积累,我们自主开发了AS分析软件:剪接位点使用变异分析(Splice sites Usage Variation Analysis,SUVA)(Cheng, Liu et al. 2021)
研究内容和结果
在SUVA的工作原理中,根据剪接位点的使用变化情况定义了五种不同类型的AS事件模型(图1a)。每种类型包含两个成对的剪接接头(SJ);SUVA通过识别这些成对的剪接接头来进行统计和分析。在SUVA的工作流中,共有5个工作模块:准备(Prepare)模块,NIR模块,IR模块,经典AS事件注释模块,以及最后的显著差异AS事件模块(图1b)。五种模块流程化分析,最终可以鉴定出不同样品组中发生显著变化的AS事件。图1c则介绍了构建四种NIR类型(alt3p、alt5p、olp、contain)的SJs集和剪接模式簇,以及到量化和差异分析的详细信息。
最让人兴奋的是,SUVA对于复杂的AS事件检出率和准确率都比其他的AS分析软件要高。复杂AS事件指的是预定义剪接模型内的一个或多个剪接位点与模型外剪接位点配对的情况,如图2a所示。对于复杂的AS事件,SUVA具有化繁为简的能力;
单细胞测序大数据亮点挖掘项目成果和案例展示
案例解析
单细胞转录组数据揭示小鼠和人的腹主动脉瘤巨噬/单核细胞的保守调控程序
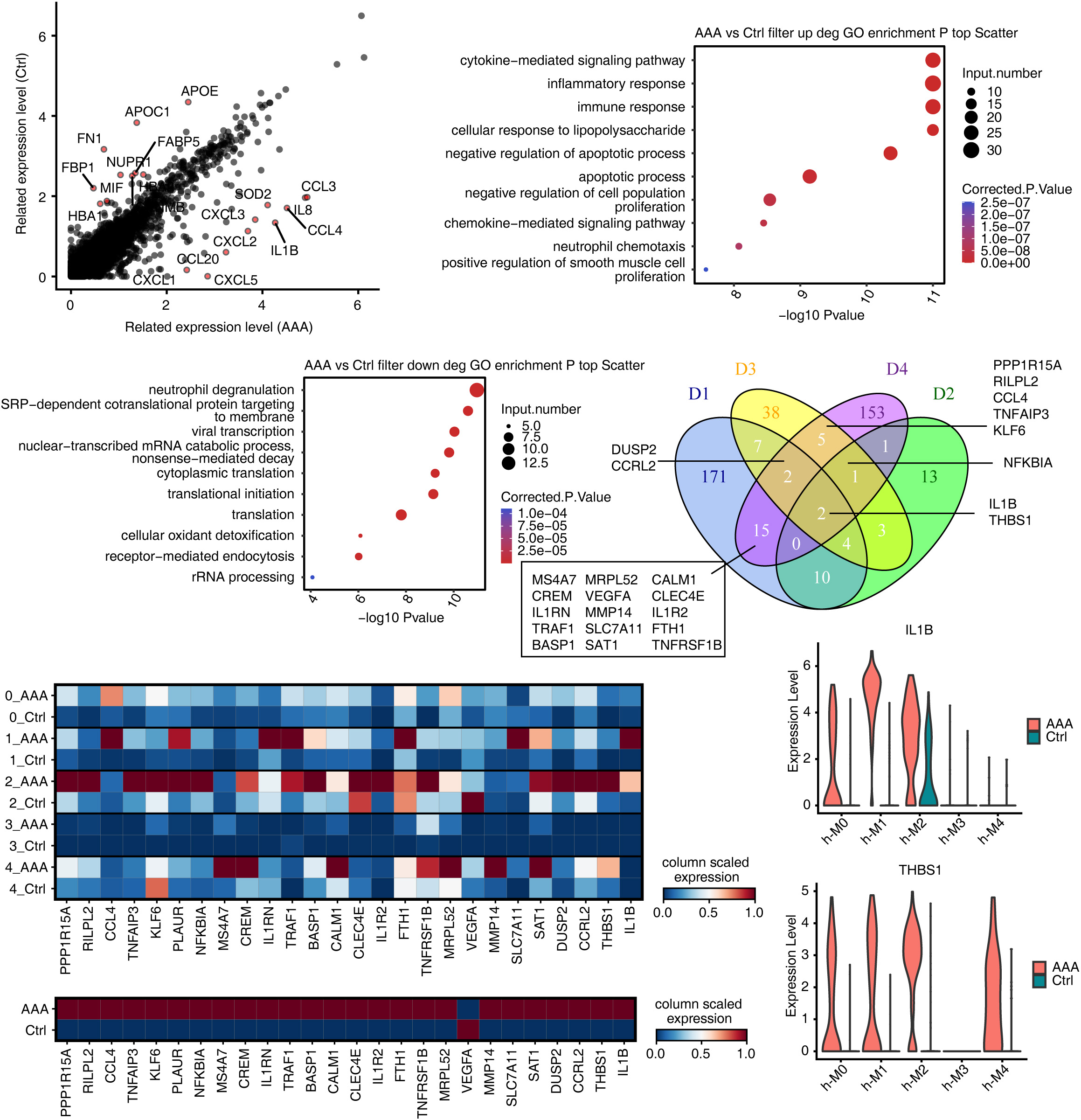
2023年1月在Frontiers in Cardiovascular Medicine期刊上发表了题为“Single-cell transcriptome in silico analysis reveals conserved regulatory programs in macrophages/monocytes of abdominal aortic aneurysm from multiple mouse models and human”。本研究基于已发表的来自不同小鼠腹主动脉瘤模型和人腹主动脉瘤组织的GSE152583、GSE164678、GSE191226和GSE166676的scRNA-seq数据集,探讨了AAA过程中巨噬/单核细胞(mφ/Mo)的细胞异质性和保守的转录调控模式,以阐明某些基因在腹主动脉瘤发病机制中的潜在关键作用。
2023年1月在Frontiers in Cardiovascular Medicine期刊上发表了题为“Single-cell transcriptome in silico analysis reveals conserved regulatory programs in macrophages/monocytes of abdominal aortic aneurysm from multiple mouse models and human”。本研究基于已发表的来自不同小鼠腹主动脉瘤模型和人腹主动脉瘤组织的GSE152583、GSE164678、GSE191226和GSE166676的scRNA-seq数据集,探讨了AAA过程中巨噬/单核细胞(mφ/Mo)的细胞异质性和保守的转录调控模式,以阐明某些基因在腹主动脉瘤发病机制中的潜在关键作用。
主要结果
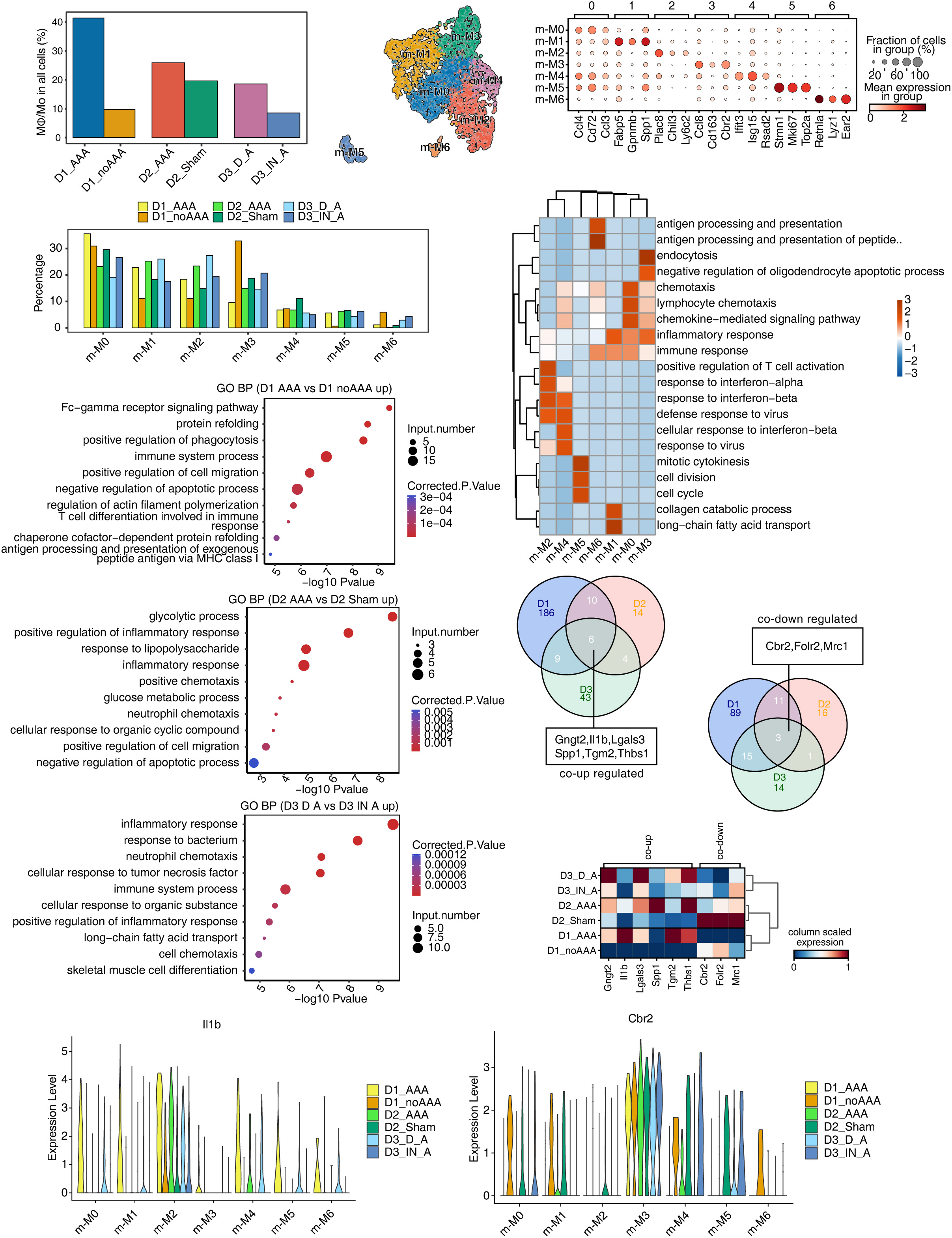
01 小鼠AAA和对照样本之间存在复杂的mφ/Mo异质性和保守的调控基因
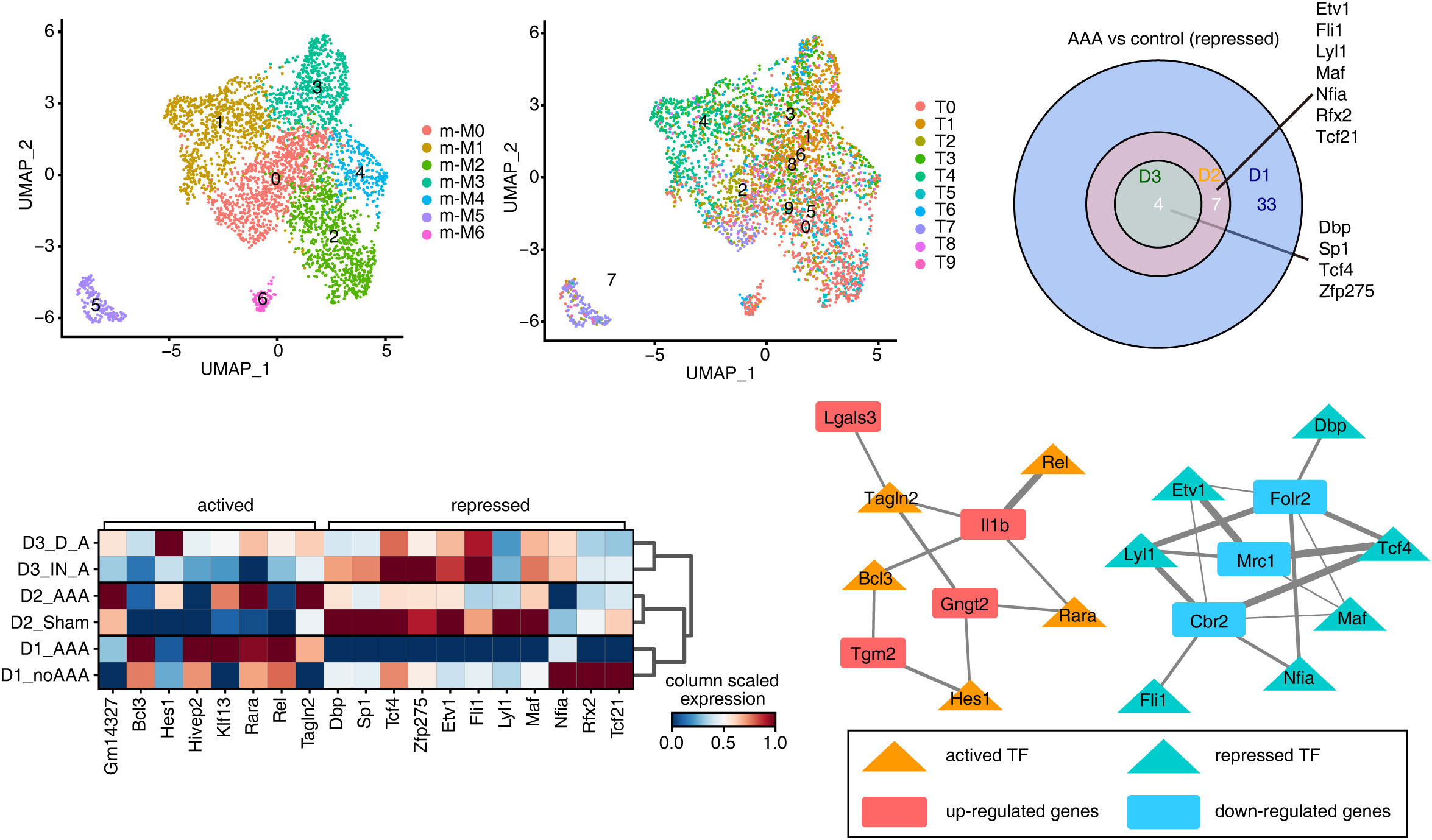
02不同AAA模型具有相似的转录因子抑制特征
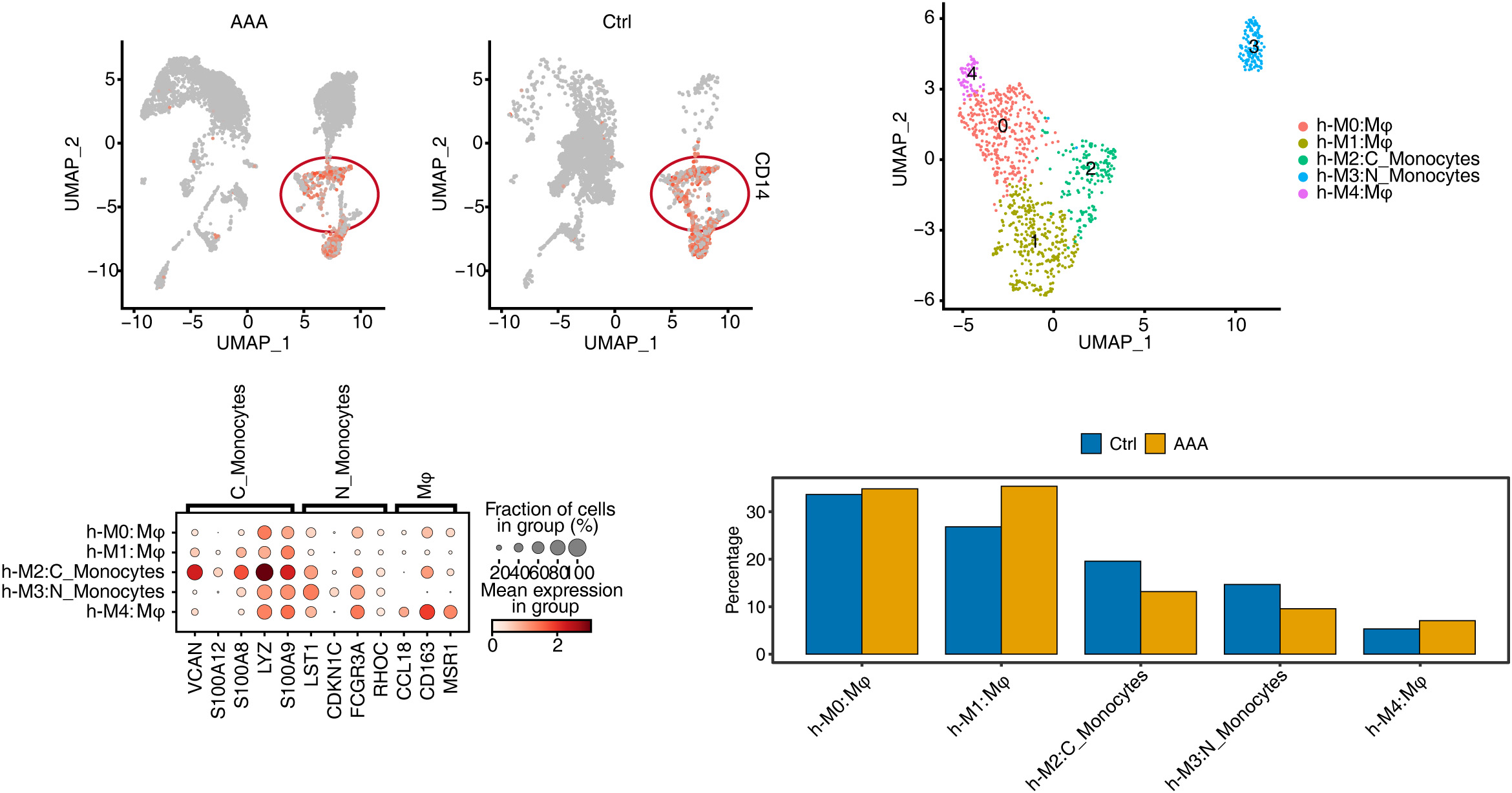
03 AAA患者的scRNA-seq显示巨噬细胞增多
04在AAA发育过程中,IL-1B和THBS1是mφ/Mo中最保守的调控基因
总结讨论
综上所述,这是第一个在单细胞水平上比较不同小鼠AAA模型中mφ/Mo基因表达调控的研究。分析显示,三种小鼠模型中mφ/Mo的co-DEGs在AAA的发展中发挥了关键作用。此外,首次分析和比较了不同小鼠AAA模型中的转录调节网络。这些共变化的转录因子与co-DEGs构成了密切相互作用的调控网络,调控巨噬细胞的内吞、增殖和凋亡。其中IL-1B和THBS1是4个数据集中共同上调的基因,说明它们在AAA形成的关键环节中起着较为保守的至关重要的作用。通过这些比较,我们可以看到当前AAA模型中的细胞分类、基因表达和转录调控网络,使我们更好地掌握了不同模型在分子水平上的异同,也为开发符合人类AAA的动物模型以及针对AAA的靶向干预提供了新的思路。