RXBio Translates Sequence to Science and Industry
Tel: 027-87050299Email: sales@rxbio.cc
RXBio Translates Sequence to Science and Industry
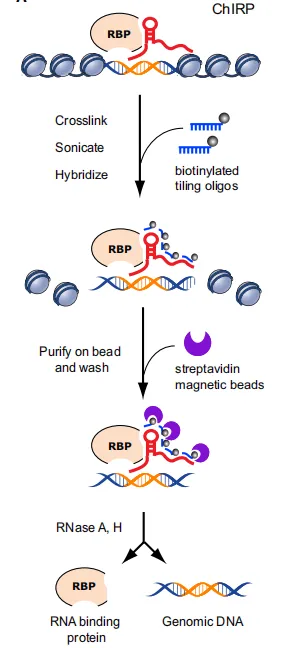
在真核细胞中,遗传信息由染色质承载,其结构和功能的持续变化对于基因表达的控制至关重要。最近的研究表明,非编码RNA(ncRNA)在染色质重组、组蛋白修饰以及转录调控等方面起着至关重要的作用。为了深入理解ncRNA与染色质之间的相互作用,科学家们开发了染色质RNA纯化(ChIRP)技术。
长链非编码RNA(lncRNA)通过与染色质形成复合体,指导表观遗传修饰或基因表达。研究显示,ChIRP技术能定位lncRNA在染色体上的结合区域,并鉴定其结合的蛋白复合物。
ChIRP技术结合测序或qPCR,用于研究lncRNA或其他RNA分子在染色质上的结合位点。研究发现,lncRNA HOTTIP通过与非编码区结合,调控miRNA基因座的转录,并影响基因表达。
ChIRP技术用于鉴定RNA在细胞核内的功能位点,解析其在基因调控、细胞分化、发育中的作用。研究发现,lncRNA NEAT1结合到调控表皮分化的基因启动子区域。
ChIRP技术在癌症、神经退行性疾病等领域研究中,用于分析与疾病相关的RNA分子如何通过染色质相互作用影响疾病进展。研究显示,lncRNA DBET和HOXDeRNA通过与特定蛋白复合物结合,影响膀胱癌和胶质瘤的进展。
✔ 不受RNA的二级结构影响:每隔100 bp设计一个探针,覆盖全长RNA分子。
✔ 分组探针设计:将所有探针分为奇数组(odd组),偶数组(even),分别进行后续下拉实验。
✔ 同时研究多种分子互作机制:同时研究LncRNA互作蛋白质、DNA和RNA。
①探针设计:设计反义寡核苷酸探针,长度20nt 左右;
②细胞培养:ChIRP实验需要的细胞数,即2×107;
③细胞甲醛交联及收集细胞;
④交联后DNA和RNA的超声打断;
⑤生物素标记的LncRNA探针与裂解液杂交;
⑥RNA、DNA及核酸结合蛋白提取,分析实验数据
英文题目:LncRNA CRLM1 inhibits apoptosis and promotes metastasis through transcriptional regulation cooperated with hnRNPK in colorectal cancer
中文标题:LncRNA CRLM1通过转录调控与hnRNPK协同作用在结直肠癌中抑制细胞凋亡并促进转移
期刊:Cell and Bioscience
影响因子:6.1

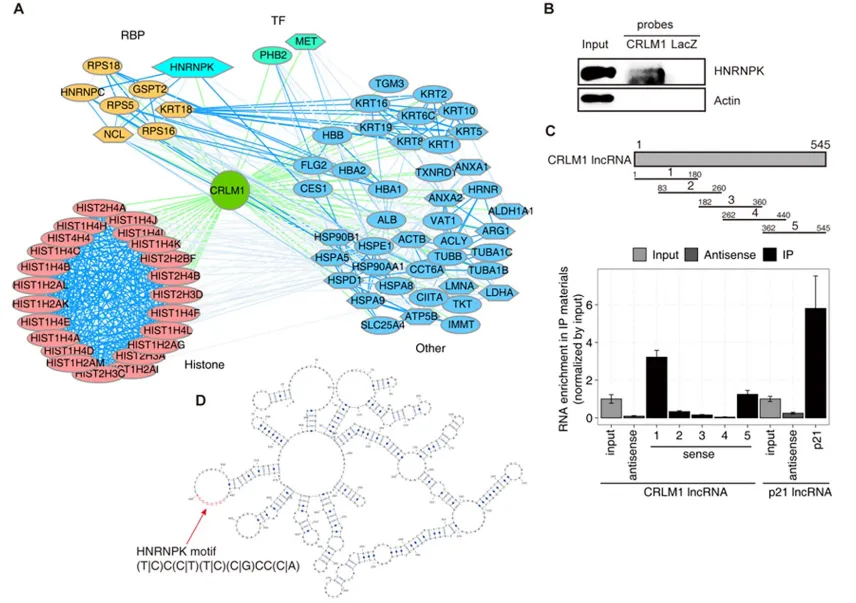
尽管结直肠肝转移(CRLM)的存活率依旧不高,但目前调节CRLM的临床指标和机构数量仍然有限。长链非编码RNA(lncRNA)作为一种新兴的细胞侵袭与转移主要调控因子,其在结直肠癌(CRC)转移中的角色和调控机制尚未完全明了。本研究通过分析公开的转录组数据,筛选并确认了与CRLM相关的lncRNAs。通过功能增减实验,我们探究了lncRNA CRLM1在体内体外的生物学功能。采用RNA测序(RNA-seq)、RNA纯化染色质分离(ChIRP)、免疫荧光(IF)、定量实时PCR(qRT-PCR)、蛋白印迹法和拯救实验等方法,深入研究了CRLM1的分子作用机制。同时,本研究还识别了与CRLM1相互作用的蛋白质、DNA和RNA。研究对lncRNA在CRLM、原发性CRC和正常组织中的表达变化进行了深入分析,揭示了一系列与转移相关的lncRNAs,其中包括CRLM1。CRLM1能够抑制Balb/C裸鼠CRC细胞的凋亡并促进肝转移。CRLM1与参与细胞粘附和DNA损伤的基因的染色质区域存在弱相关性,这种相关性与CRLM1调控的前转移基因表达呈双向相关。CRLM1与hnRNPK蛋白发生物理相互作用,并促进其核定位。CRLM1显著提高了hnRNPK启动子的占有率,并共同调控了一组转移基因的表达。
CRLM1显示出与涉及细胞粘附和DNA损伤的基因的染色质区域的微弱联系,这种联系与CRLM1所调控的前转移基因表达呈双向关联。CRLM1与hnRNPK蛋白存在物理上的相互作用,并有助于其在细胞核内的定位。此外,CRLM1显著提升了HNRNPK启动子的占有率,并协同调控了一组转移基因的表达。
英文题目:The long non-coding RNA PFI protects against pulmonary fibrosis by interacting with splicing regulator SRSF1
中文标题:长链非编码 RNA PFI 通过与剪接调节因子 SRSF1 相互作用来预防肺纤维化
期刊:Cell Death and Differentiation
影响因子:13.7

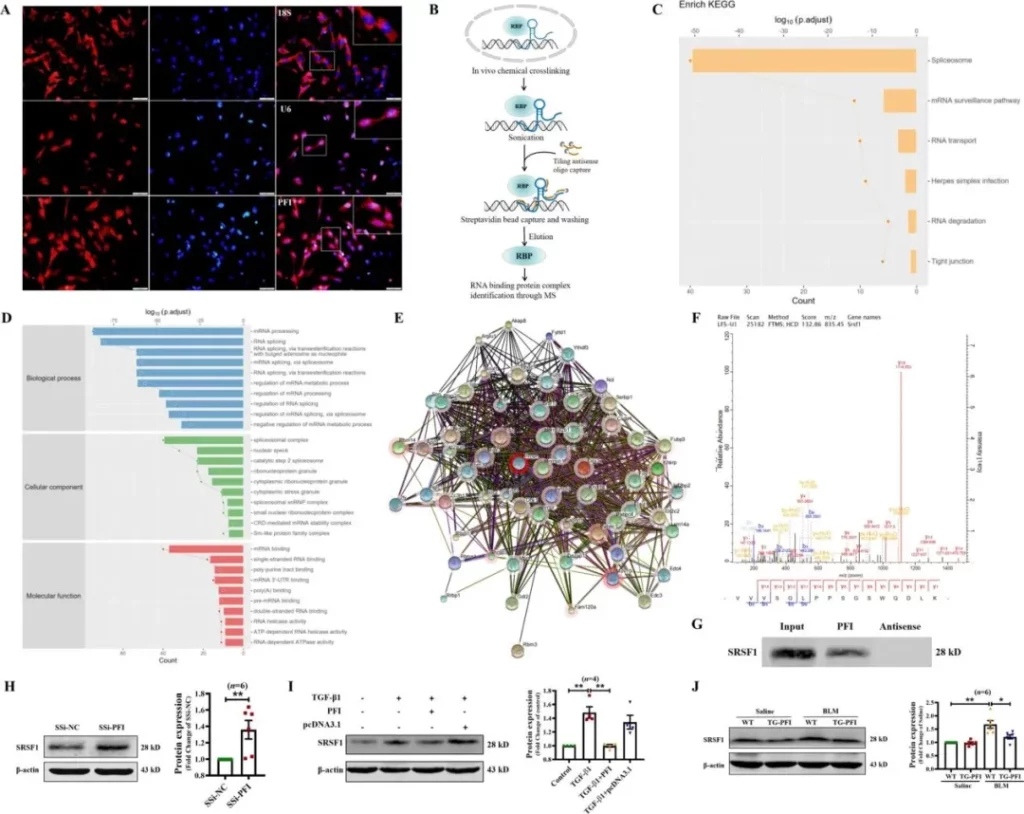
肺纤维化,亦称Pulmonary fibrosis (PF),是一种死亡率高且病因复杂的间质性肺炎。其特征在于肺泡间质的逐渐瘢痕化和肌成纤维细胞的病变。近年来,长链非编码RNA (long non-coding RNAs, lncRNAs)在器官纤维化中的作用日益受到重视。本项研究主要分析了lncRNA在肺纤维化中的作用机制。
利用ChIRP技术和RNA pull down实验,成功分离出与目标lncRNA相互作用的蛋白质。研究发现,被本研究定义为肺纤维化抑制剂PFI的lncRNA-NONMMUT060091,能够直接与富含丝氨酸/精氨酸的剪接因子1(SRSF1)结合,并抑制其表达及其促进纤维化的活性。研究数据显示,PFI通过下调SRSF1的表达和活性,减少EDA+Fn1剪接异构体的产生,进而减轻肺纤维化症状。这表明PFI和SRSF1可能是治疗肺纤维化的潜在靶点。这项研究表明lncRNA-PFI的抗纤维化功能在肺纤维化中具有重要作用。运用ChIRP-MS和RNA pull down检测技术,发现lncRNA PFI通过结合并抑制剪接因子SRSF1的表达和活性,抑制EDA + Fn1剪接异构体的产生,从而参与肺纤维化的调控。因此可以推测,诱导PFI表达可能为预防和治疗PF提供一种新的途径。
英文题目:N4-Acetylcytidine-Mediated CD2BP2-DT Drives YBX1 Phase Separation to Stabilize CDK1 and Promote Breast Cancer Progression
中文标题:N4-乙酰胞苷介导的 CD2BP2-DT 驱动 YBX1 相分离以稳定 CDK1 并促进乳腺癌进展
期刊:Advanced Science
影响因子:14.3

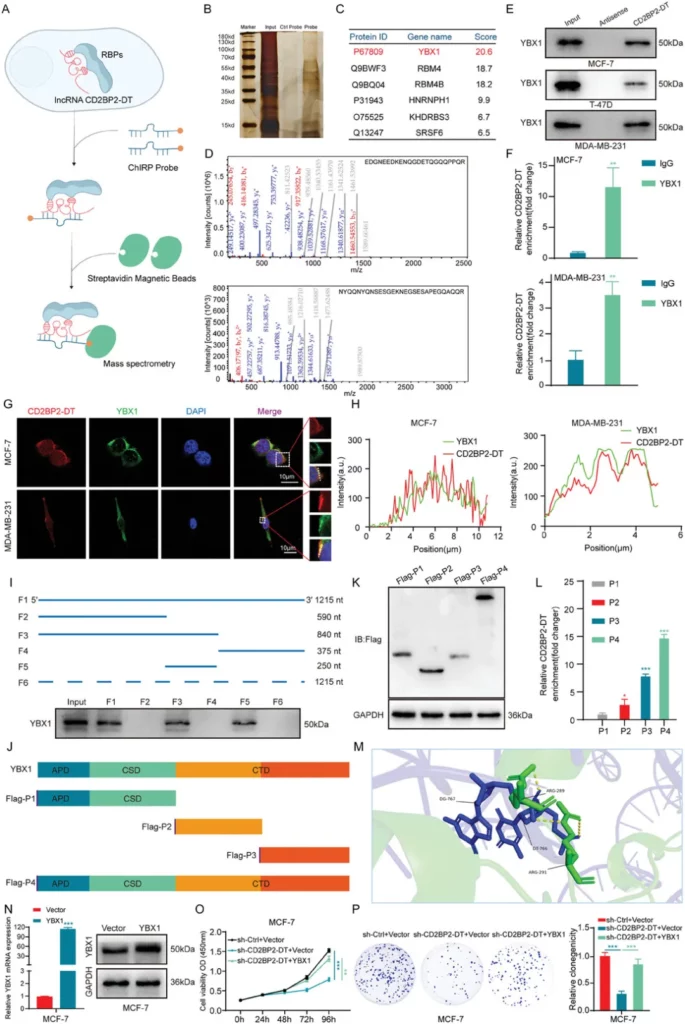
在乳腺癌的形成与进展过程中,长链非编码RNA(lncRNAs)扮演着至关重要的角色。尽管如此,lncRNA在乳腺癌中的确切作用和生物学功能仍旧不明确。通过生物信息学的研究,科学家们发现了一种新的lncRNA,名为CD2BP2-DT,它在乳腺癌组织中表达量较高,并且与较差的临床结果和较低的总生存率紧密相关。实验结果表明,CD2BP2-DT能够促进乳腺癌细胞的增殖。进一步研究揭示,NAT10参与了CD2BP2-DT的n4-乙酰胞苷(ac4C)修饰过程,这一修饰增强了RNA的稳定性和表达水平。更为关键的是,CD2BP2-DT通过促进YBX1相分离,进而增强了CDK1 mRNA的稳定性,从而推动了乳腺癌细胞的增殖。综上所述,lncRNA CD2BP2-DT被确认为乳腺癌细胞增殖的关键调节因子,其作为乳腺癌潜在生物标志物和治疗靶点的潜力备受关注。
研究表明,lncRNA-RBP相互作用在癌症发展中至关重要。通过ChIRP实验和质谱分析,我们发现CD2BP2-DT与特定蛋白质有相互作用。YBX1被选为CD2BP2-DT的结合伙伴,它是一种多功能癌蛋白,与乳腺癌进展密切相关。Kaplan-Meier分析显示YBX1高表达与不良预后相关。生物素标记的RNA拉下和RIP实验确认了CD2BP2-DT和YBX1在乳腺癌细胞中的相互作用。共定位分析显示它们主要共定位在细胞质中。利用RNAfold软件预测CD2BP2-DT的二级结构,并通过缺失定位分析确定了与YBX1相互作用的关键区域。构建截断YBX1蛋白的实验揭示了参与相互作用的YBX1结构域。分子对接模拟进一步分析了CD2BP2-DT与YBX1的结合位点。
参考文献
Wang, Z., Chen, J., Sun, F. et al. LncRNA CRLM1 inhibits apoptosis and promotes metastasis through transcriptional regulation cooperated with hnRNPK in colorectal cancer. Cell Biosci 12, 120 (2022).
Sun, J., Jin, T., Su, W. et al. The long non-coding RNA PFI protects against pulmonary fibrosis by interacting with splicing regulator SRSF1. Cell Death Differ 28, 2916–2930 (2021).
H. Wang, B. Zhao, J. Zhang, Q. Hu, L. Zhou, Y. Zhang, Y. Cai, Y. Qu, T. Jiang, D. Zhang, N4-Acetylcytidine-Mediated CD2BP2-DT Drives YBX1 Phase Separation to Stabilize CDK1 and Promote Breast Cancer Progression. Adv. Sci. 2025, 12, 2411834.

电话:027-870502099
邮箱:sales@rxbio.cc
地址:武汉市东湖高新区高新二路388 号
光谷生物医药加速器 18 栋 1-2层
单细胞多组学 空间转录组
三代测序 功能基因组
表观遗传学 互作组学
单细胞大数据 数据深度挖掘

欢迎关注公众号「瑞兴生物」