RXBio Translates Sequence to Science and Industry
Tel: 027-87050299Email: sales@rxbio.cc
RXBio Translates Sequence to Science and Industry
二代转录组测序不能提供一个基因多个转录本分别定量的结果,因为该技术并不适用于这种情况。二代短测序单条read无法跨越全长转录本,需要借助软件将短reads进行拼接以得到转录本,然后再评估表达量,这可能存在拼接错误或不完整的问题,无法准确获得完整转录本,并且不能区分一个基因多个转录本reads的来源,不同基因相似高度保守区短reads存在多比对现象,导致转录本甚至基因水平表达定量不准确或出现偏倚。而Nanopore三代全长测序无需打断和拼接转录本,一条read可跨越全长转录本,多比对率低,能够准确获取一个基因多个全长转录本各自的表达信息。 从二代转录组到nanopore三代全长转录组,实现了平台升级和技术革新,能够解决二代测序无法解决的问题。
然而,相较于二代测序技术,三代测序技术的每条reads长度显著更长。在测序数据量相同的情况下,三代测序所产生的reads数量会明显少于二代测序。对于某些表达量较低的转录本而言,这就要求达到极高的测序深度才能实现有效检测。例如,在进行人类转录组的饱和检测时,需要超过30Gb的测序数据量。如此高的要求必然会大幅增加第三代测序的成本,给研究工作带来更大的经济负担。靶向测序技术能够让研究人员筛选出感兴趣的基因位点,从而降低测序成本和工作量,以便在目标区域获取高覆盖率的数据。
ONT靶向长读长测序(Oxford Nanopore Technologies Targeted Long-Read Sequencing,ONT-TlrRNA-seq)是通过PCR扩增感兴趣的转录本全长区域,随后直接对扩增子进行测序。
ONT-TlrRNA-seq技术的核心优势在于,它能够针对特定的转录本进行深度测序,而无需对整个转录组进行全面测序。这种方法不仅显著提升了测序效率,还使研究人员能够更加专注于与疾病或特定生物学过程直接相关的转录本。
通过ONT-TlrRNA-seq技术,研究人员可以快速识别关键基因的转录本表达情况,为后续的转录本功能研究和疾病诊断提供有力支撑。此外,ONT-TlrRNA-seq技术还因其灵活性高、操作简便而广受青睐,成为当前转录本检测领域的一项重要技术。
✔ 测序长度数千至上万bp,覆盖全长。
✔ 无需拼接,天然全长。
✔ 可直接识别转录本isoform。
✔ 精准定量,低表达转录本亦可检测。
✔ 测序量低,性价比高。

• 提取的RNA样本:总量≥5 ug,浓度≥100 ng/uL,纯度:260/280 ≥1.8、细胞样本:≥1×107cells
• 新鲜或冻存组织样本:≥200 mg
• 全血:≥ 4 mL
案例1
文献1: Patients with Asian-type DEL can safely be transfused with RhD-positive blood
中文标题:亚洲型DEL患者可以安全地使用RhD阳性血液输血
期刊:Blood
影响因子:21
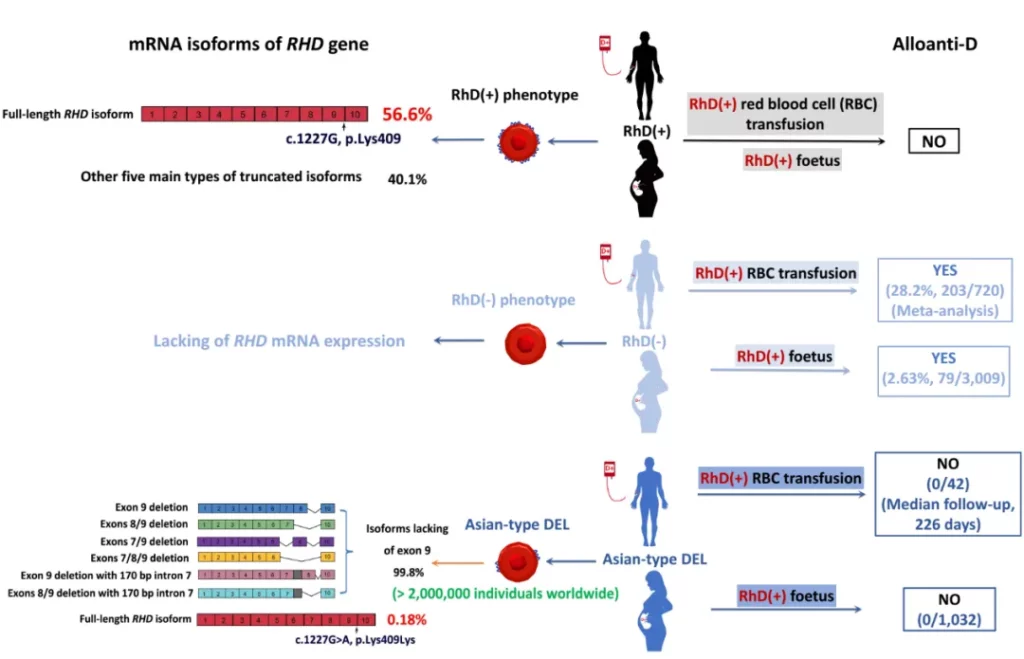
该研究从三个方面为“亚洲型”DEL血型患者安全输注RhD阳性血液提供了有力证据。
首先,首次开展了针对“亚洲型”DEL血型患者输注RhD阳性红细胞的多中心临床试验。通过长期随访发现,“亚洲型”DEL血型患者能够安全输注RhD阳性红细胞,且未出现抗 – D同种免疫现象。
其次,开展了全国范围内“亚洲型”DEL血型孕妇的回顾性研究。大样本数据证实,“亚洲型”DEL血型孕妇在怀有RhD阳性胎儿时,不会发生抗 – D同种免疫,这与RhD真阴性“熊猫血”孕妇存在统计学差异。同时,在已产生同种抗D的孕妇中,也未发现“亚洲型”DEL血型孕妇。
最后,在“亚洲型”DEL血型个体的有核红细胞中,借助Nanopore三代测序技术,首次检测出携带有1227A突变的RHD基因全长转录本。体外实验证实,其能够表达具有完整表位的RhD抗原,与RhD阳性红细胞抗原表位一致。这表明该血型患者可能通过免疫耐受来避免同种免疫的发生。
上述研究成果,为制定“亚洲型”DEL血型患者常规输注普通RhD阳性血液的临床输血规范提供了极为有力的证据,进而能够解决该血型患者血液供应困难的问题。
此外,“亚洲型”DEL血型的孕妇无需注射抗D免疫球蛋白,也不必频繁进行孕期抗D同种免疫监测。这不仅能节省抗D免疫球蛋白这一珍贵的血液制品,还可为这类血型的孕妇节省每年数千万的抗D免疫球蛋白注射及监测费用。此研究成果还能让日本、韩国、东南亚国家以及有众多东亚移民的发达国家中的“亚洲型”DEL血型群体从中受益。
案例2
文献2: Discovery of circular transcripts of the human BCL2-like 12 (BCL2L12) apoptosis-related gene, using targeted nanopore sequencing, provides new insights into circular RNA biology
中文标题:通过靶向纳米孔测序技术发现人类BCL2样12(BCL2L12)凋亡相关基因的环状转录本,为环状RNA生物学提供了新的见解
期刊:Funct Integr Genomics
影响因子:3.9
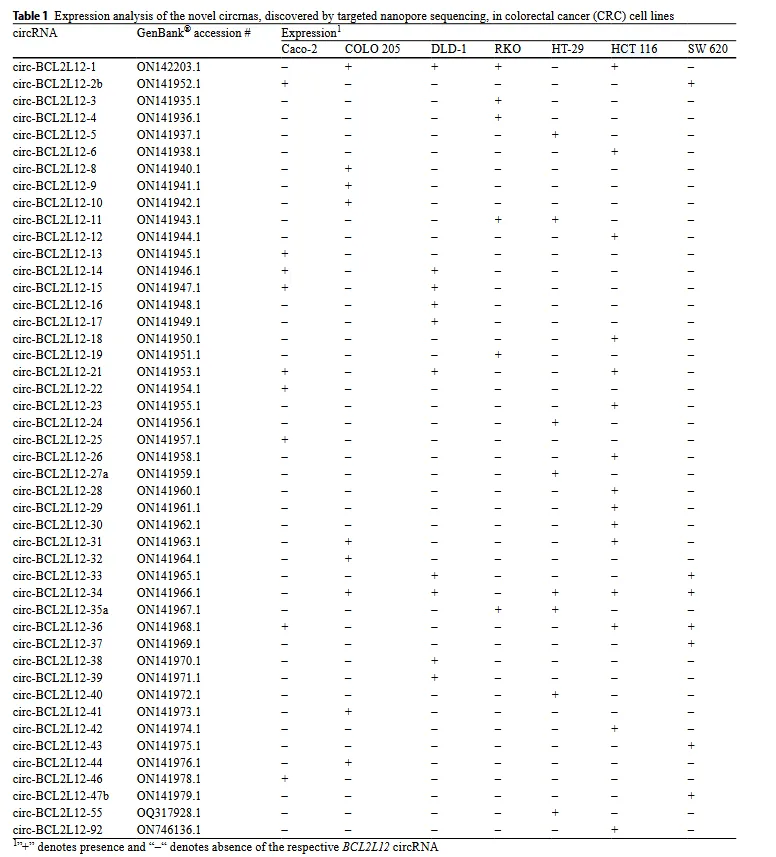
基于靶向纳米孔测序所发现的新型环状RNA(circRNAs)在结直肠癌(CRC)细胞系中的表达分析
研究人员设计了 2 对针对 BCL2L12 每个注释外显子的发散引物,开展嵌套 PCR。把不同细胞系的 PCR 产物混合纯化后构建纳米孔测序文库,接着利用公开工具和自研算法开展生物信息学分析,成功研发出一种识别新型 circRNAs 的新流程。在 CRC 细胞系里,研究人员一共鉴定出 46 种 BCL2L12 circRNAs,其中大部分是首次发现。这些 circRNAs 覆盖了 BCL2L12 基因约一半的区域,并且和已有的公共数据库里记录的 circRNAs 相比,仅有少量重叠。
另外,部分 circRNAs 由单个外显子构成,还有一些包含从未描述过的外显子,这些外显子的频率和覆盖度呈现出独特特征。通过纳米孔测序数据分析,研究人员发现 BCL2L12 circRNAs 在 7 种 CRC 细胞系中的表达有显著差异。不同细胞系中 circRNAs 的外显子结构和内含子覆盖状况各不相同,其中外显子 4 和 5 在 circRNAs 中出现的频率较高,不过它们在不同细胞系 circRNAs 中的具体结构存在差别。
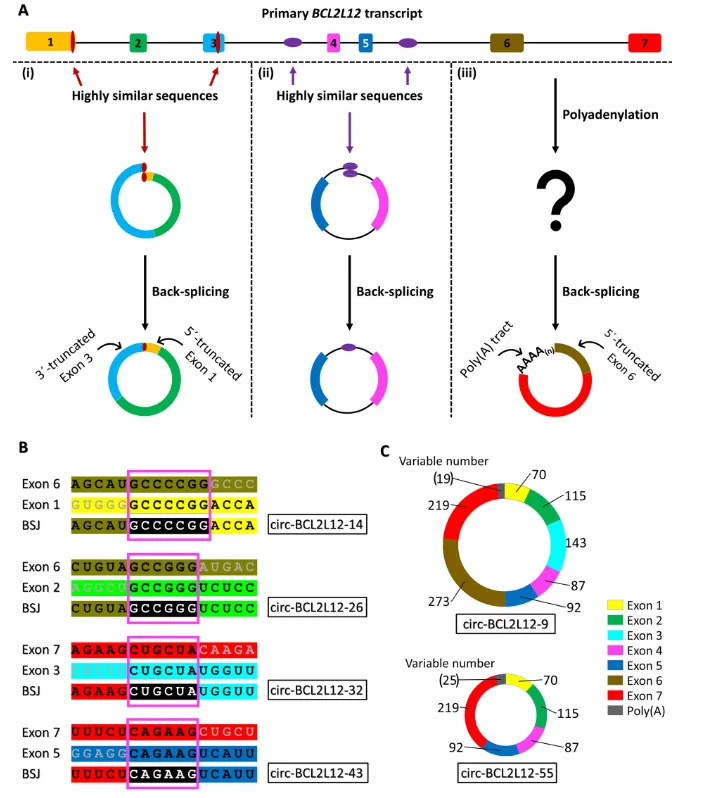
在鉴定出的 46 种 circRNAs 中,研究人员观察到 40 种不同的反向剪接位点(BSJ)。大部分 circRNAs 的反向剪接位点不符合规范,并且在多数情形下,形成 BSJ 的前体 mRNA 区域具备高度的序列相似性。此外,研究还发现 2 种 circRNAs(circ – BCL2L12 – 9 和 circ – BCL2L12 – 55)含有类似于信使核糖核酸(mRNA)多聚腺苷酸尾(poly (A) tail)的多聚腺苷酸(poly (A))片段,这一发现对传统的 circRNAs 认知提出了挑战。
1.Ji, Y.,Luo, Y.,Wen, J.,Sun, Y.,Jia, S.,Ou, C.,Yang, W.,Chen, J.,Ye, H.,Liu, X.,Liang, Y.,Lu, Z.,Feng, Y.,Wu, X.,Xiao, M.,Mo, J.,Zhou, Z.,Wang, Z.,Liao, Z.,Chen, J.,Wei, L.,Luo, G.,Santoso, S.,Fichou, Y.,Flegel, W.A,Shao, C.,Li, C.,Zhang, R., & Fu, Y. (2023). Patients with Asian-type DEL can safely be transfused with RhD-positive blood. Blood, 141 (17), 2141-2150.
https://doi.org/10.1182/blood.2022018152
2.Karousi, P., Kontos, C.K., Nikou, S.T. et al. Discovery of circular transcripts of the human BCL2-like 12 (BCL2L12) apoptosis-related gene, using targeted nanopore sequencing, provides new insights into circular RNA biology. Funct Integr Genomics 25, 66 (2025). https://doi.org/10.1007/s10142-025-01578-1

电话:027-870502099
邮箱:sales@rxbio.cc
地址:武汉市东湖高新区高新二路388 号
光谷生物医药加速器 18 栋 1-2层
单细胞多组学 空间转录组
三代测序 功能基因组
表观遗传学 互作组学
单细胞大数据 数据深度挖掘

欢迎关注公众号「瑞兴生物」