在肿瘤研究不断走向精细化的当下,单细胞三代长读长测序正成为新的突破口。近期一项发表于 Protein & Cell(IF 12.8) 的研究,仅依托单细胞三代测序数据,便构建出迄今最全面的结直肠癌全长转录本图谱,揭示了传统短读长无法捕捉的 UTR 重塑、剪接异常和 isoform 切换 等深层分子变化。
文章几乎完全依赖测序与数据分析,即实现高分发表。这从侧面反映出:全长转录本层面的信息量足够大、独特且具有新颖性,而现阶段能在单细胞水平获取这类信息的技术,只有三代长读长。
对于希望寻找新靶点、发现难以从基因表达层面捕获的机制、或提升研究创新度发表高分成果的您来说,单细胞三代测序正处于技术红利期 —— 越早利用,越能在研究产出与成果竞争中占领先机。

● 英文题目: Systematic characterization of full-length RNA isoforms in human colorectal cancer at single-cell resolution
● 中文题目: 单细胞水平解析结直肠癌全长RNA异构体的系统性特征
● 发表期刊: Protein & Cell (Q1 IF =12.8)
● 发表时间: 2025年3月
研究背景
● 基因表达并非全部
许多关键的肿瘤特征并不会体现在基因表达量上,而是隐藏在更深层的 RNA 剪接、UTR 长度变化、转录本结构重塑 中。
● 短读长技术存在天然盲区
传统二代测序只能看到 RNA 片段,无法准确重建全长 RNA,常导致 剪接事件缺失、UTR 判断不准、蛋白结构变化被忽略。
● 肿瘤异质性需要“单细胞 + 全长”
肿瘤中不同细胞可能使用完全不同的 isoform,仅靠 bulk 或短读长单细胞测序无法真实呈现。
● 单细胞三代全长转录组带来关键突破
单细胞三代测序可一次读出完整 RNA 分子,直接解析每个细胞的转录本结构,解决过去难以跨越的技术限制。
● 本研究据此构建结直肠癌全长转录组图谱
揭示传统基因表达分析无法观察的剪接调控、UTR 改变、isoform 切换与蛋白结构变化,从而阐明影响肿瘤发生、亚型分化与潜在治疗靶点的关键机制。
研究思路
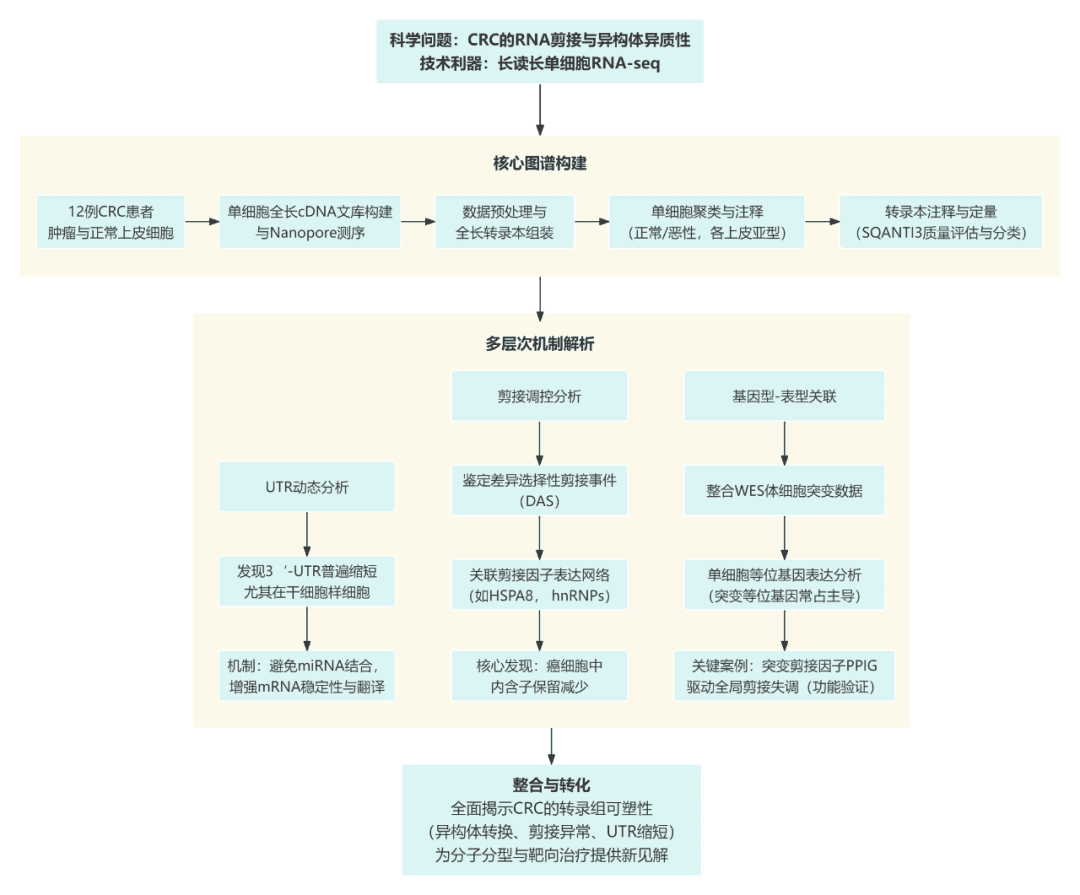
研究团队采用 SCAN-seq 与 SCAN-seq2 两种长读长单细胞测序技术,对 12 位结直肠癌患者的肿瘤与邻近正常组织进行测序。通过严格的 QC、比对与转录本拼装,获得高质量全长 RNA 序列,并对细胞进行聚类,区分正常上皮与癌细胞亚群。
第 2 阶段:解析 UTR 与剪接层面的结构变化
在构建图谱基础上,研究进一步量化基因的 3’UTR/5’UTR 长度变化,发现癌细胞尤其是 stem/TA-like 细胞出现大规模 UTR 缩短,提示 mRNA 稳定性和翻译调控被重新塑造。
同时,作者系统分析了 7 类主要剪接事件(SE、RI、A5、A3、AF、AL、MX) 的差异,识别了跨患者共享的异常剪接,并通过相关性分析揭示剪接因子与特定 AS 事件的潜在调控关系。
第 3 阶段:探究 isoform 与编码序列(CDS)层面的功能后果
作者开展 差异转录本使用(DTU) 和 差异编码序列使用(DCU) 分析,重点评估 isoform 切换是否改变蛋白结构。结果显示大量基因虽无表达量差异,但其主要 isoform 在癌细胞中发生“重写”,导致蛋白结构域变化,可能改变其致癌活性。
例如 THAP4、MYC、EWSR1 等关键基因均展示显著 isoform 选择性表达,提示 功能性剪接重塑是癌细胞的重要策略。
第 4 阶段:联合 iCMS 亚型与突变信息深化机制理解
研究进一步将全长转录组信息与结直肠癌 iCMS2/iCMS3 亚型分层结合,揭示不同亚型存在特异的剪接调控与 isoform 特征。同时利用单细胞长读长测序的优势,进行 等位基因特异性表达(ASE) 与突变转录本分析,阐明基因突变与剪接异常之间的关联。
研究发现
研究利用 SCAN-seq 与 SCAN-seq2 对多位患者的肿瘤与配对正常组织进行单细胞三代测序,获得大规模高质量 全长 RNA 分子。通过细胞聚类与注释,成功区分出正常上皮、不同癌细胞亚群及免疫/基质细胞,构建了目前最系统的 结直肠癌单细胞全长转录本图谱。与正常细胞相比,癌细胞展现出更丰富的转录本数量、更高的剪接活性与更复杂的 isoform 结构。
2、发现大规模 UTR 重塑及其亚群偏好性
研究系统分析了 3’UTR 与 5’UTR 的长度变化,发现 3’UTR 普遍缩短 是癌细胞的重要特征,且在 stem-like 与 TA-like 癌细胞中最为突出。UTR 缩短减少 miRNA 结合位点,使 mRNA 更稳定、更易翻译,提示 肿瘤通过 UTR 重构主动提升表达优势。这一现象跨患者共享,是结直肠癌中稳定存在的深层调控模式。
3、剪接异常在癌细胞中普遍存在并呈现特定模式
基于全长测序准确识别的 7 类剪接事件(如 SE、RI、A5/A3 等),研究发现癌细胞存在广泛的 剪接增多与异常选择性剪接。多种异常在不同患者间保持一致,提示其具有共同驱动意义。同时,剪接因子(SFs)在癌细胞中显著上调,与特定 AS 事件呈高度相关关系,表明剪接程序被系统性重写。
4、关键基因出现显著 isoform 切换,功能性影响深远
通过差异转录本使用(DTU)与差异编码序列使用(DCU)分析,发现大量基因虽然整体表达量不变,但主导 isoform 在癌细胞中彻底“换代”。这种 isoform 切换 会导致蛋白结构域发生丢失、延长或重排,从而改变蛋白功能。例如 THAP4、EWSR1、MYC 等关键基因在癌细胞中出现特异性致癌 isoform,提示转录本层级比基因表达更能反映癌细胞的真实状态。
5、不同结直肠癌亚型存在特异性转录本特征
将全长转录组数据与 iCMS2/iCMS3 亚型信息结合后,研究揭示不同亚型具有独特的剪接模式、UTR 结构与 isoform 组合。iCMS3(炎症主导型)细胞呈现更明显的剪接重塑与 isoform 多样性,而 iCMS2(增殖主导型)则偏向结构保守型 isoform 表达。
此外,借助长读长测序可直接观察 突变等位基因特异性表达(ASE) 与其对应的突变转录本,进一步阐明突变与剪接异常之间的联系。
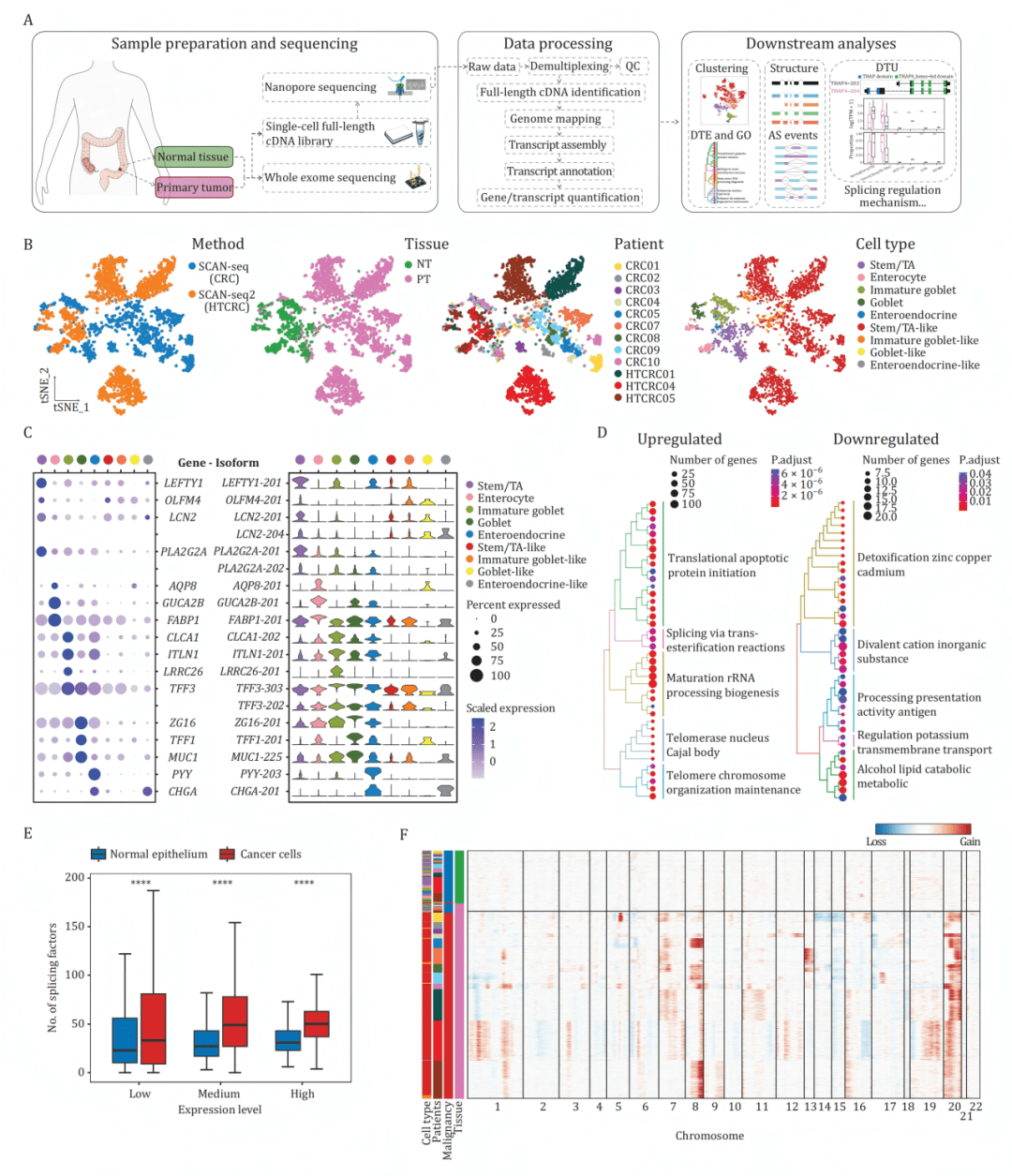
B:t-SNE图显示细胞按测序方法、组织类型(癌/正常)、患者及细胞类群清晰分离。
C:点图与小提琴图展示经典标记基因及其特异异构体在各细胞类群中的表达,证实异构体水平的分辨率更高。
D:癌症细胞中上/下调异构体对应基因的GO富集分析,显示剪接相关通路被激活。
E:癌细胞中高表达的剪接因子数量显著多于正常细胞。
F:推断的细胞染色体大规模拷贝数变异热图,显示患者间异质性。
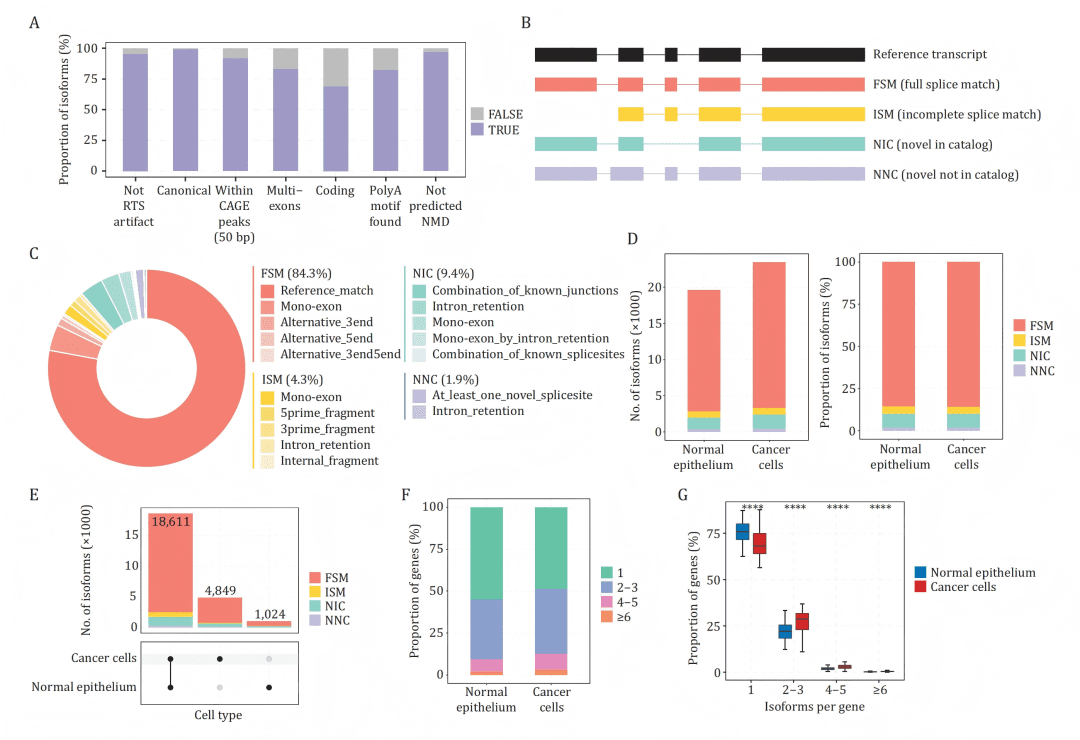
B-C:依据剪接位点与参考转录组的匹配度,将异构体分为FSM、ISM、NIC、NNC四类及其比例。
D:癌细胞表达的异构体与基因总数均高于正常上皮细胞。
E:UpSet图展示癌与正常细胞共享及特有的异构体数量。
F-G:癌细胞中表达多个异构体的基因比例更高,且主要表达集中在1-2个主导异构体上。
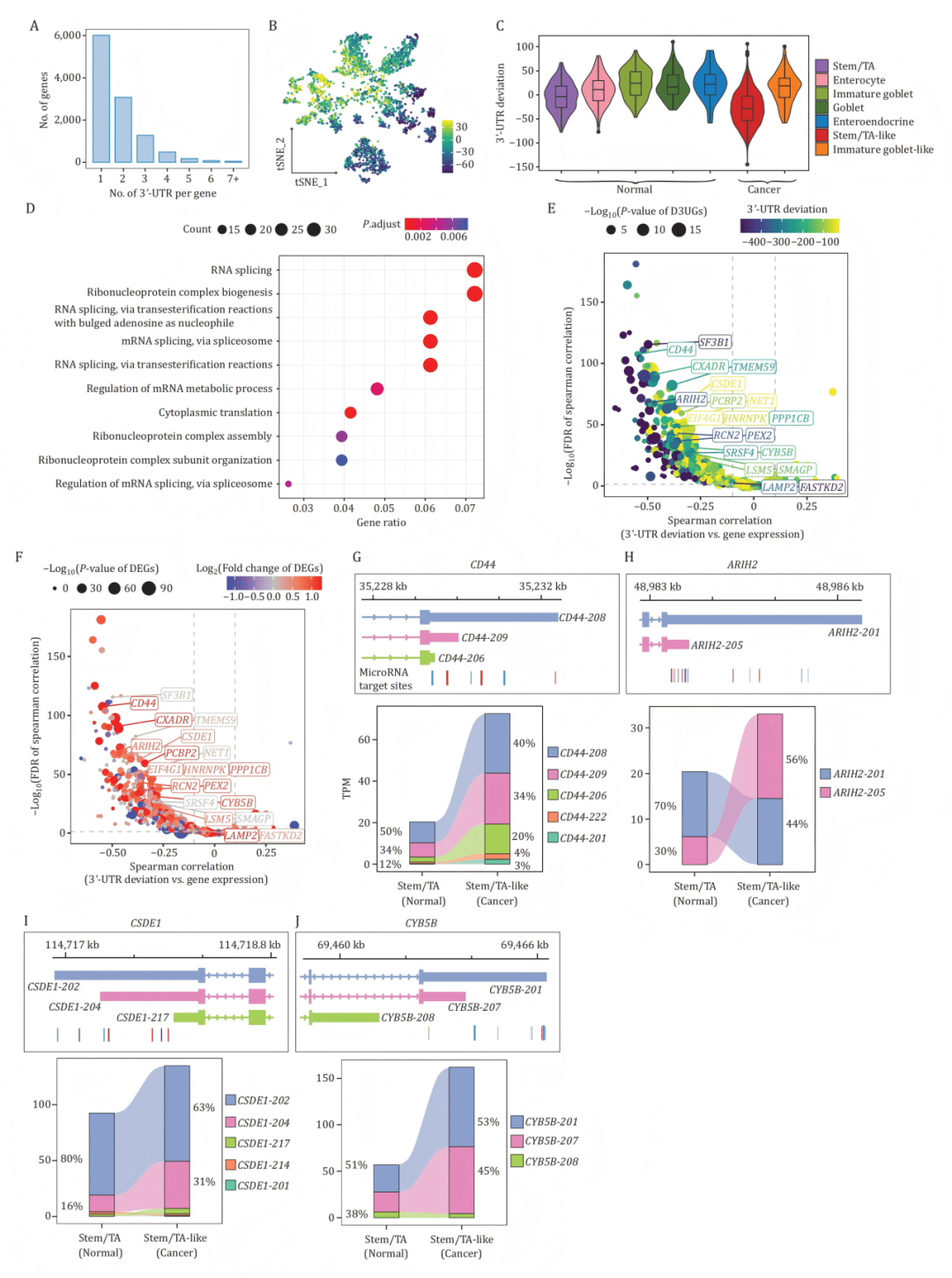
B-C:t-SNE图及小提琴图显示癌细胞(尤其是干细胞样细胞)普遍存在3‘-UTR缩短。
D:3‘-UTR缩短基因富集于RNA剪接等通路。
E-F:火山图显示3‘-UTR缩短与对应基因表达上调显著相关。
G-J:以CD44、ARIH2等基因为例,展示其缩短的3‘-UTR异构体结构(失去部分miRNA结合位点)及其在癌细胞中表达比例上升。
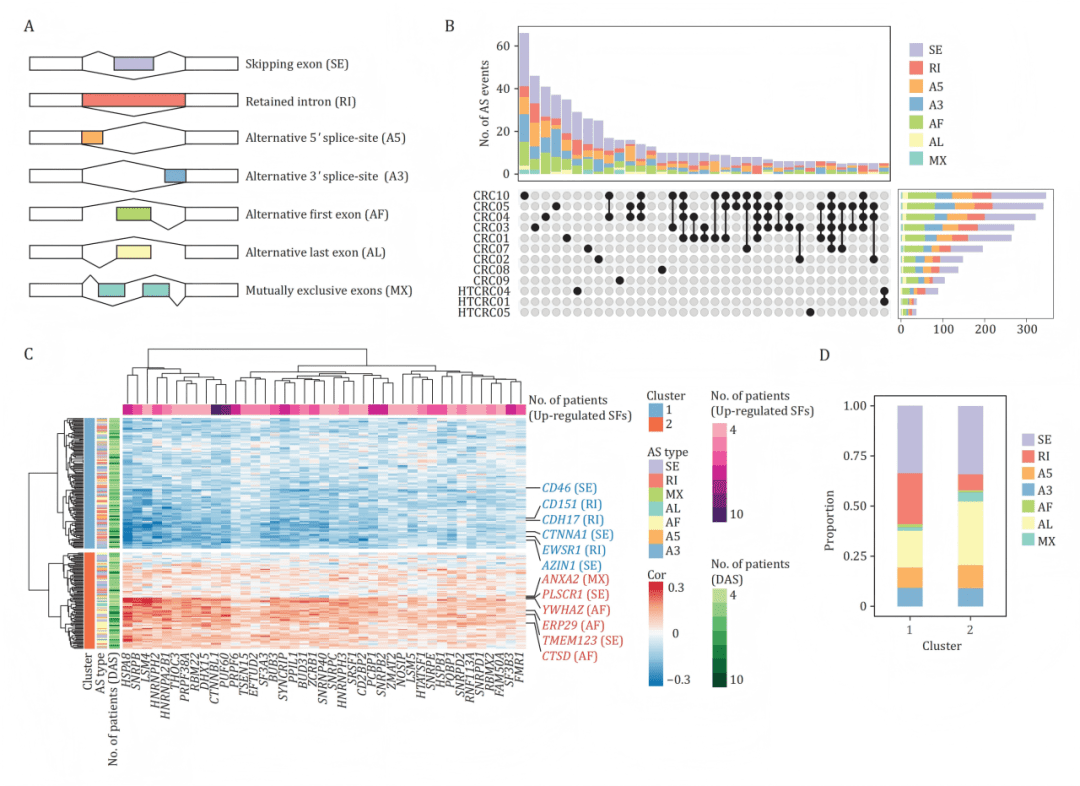
B:UpSet图显示差异剪接事件在不同患者间既有共享也存在异质性。
C:热图展示差异剪接事件PSI值与差异表达剪接因子表达量的相关性,聚类为两个正负相关簇。
D:Cluster 1富含内含子保留事件,且与剪接因子表达负相关;Cluster 2则相反。
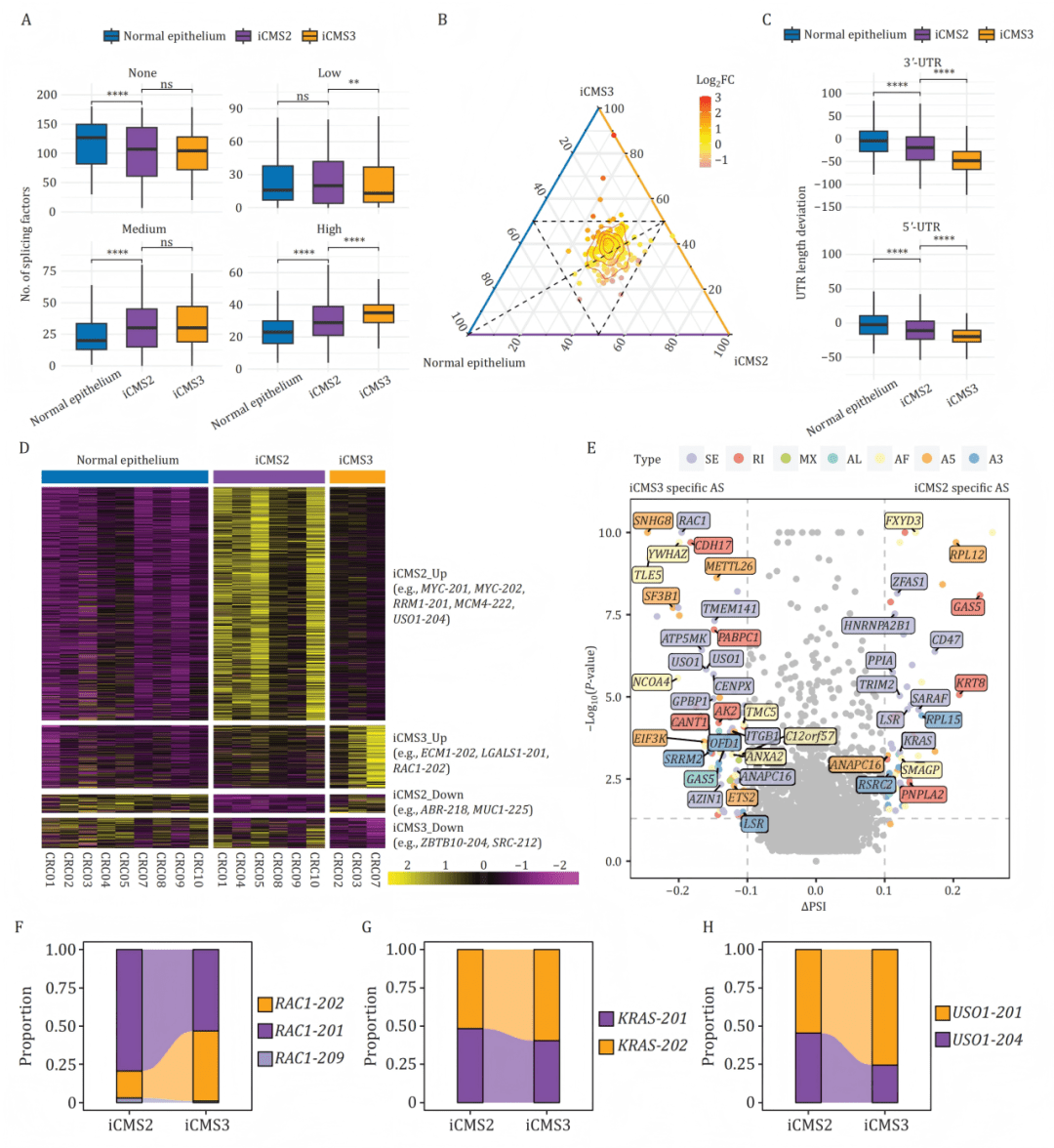
B:三元图展示剪接因子在正常、iCMS2、iCMS3细胞中的表达分布模式。
C:iCMS3亚型癌细胞表现出最显著的3‘-UTR和5‘-UTR缩短。
D:热图展示各亚型特异上调的异构体(如iCMS2的MYC异构体,iCMS3的RAC1b异构体)。
E-H:火山图及比例堆叠条形图显示RAC1、KRAS等基因的异构体使用在亚型间存在特异性差异(如iCMS3中促迁移的RAC1b比例更高)。
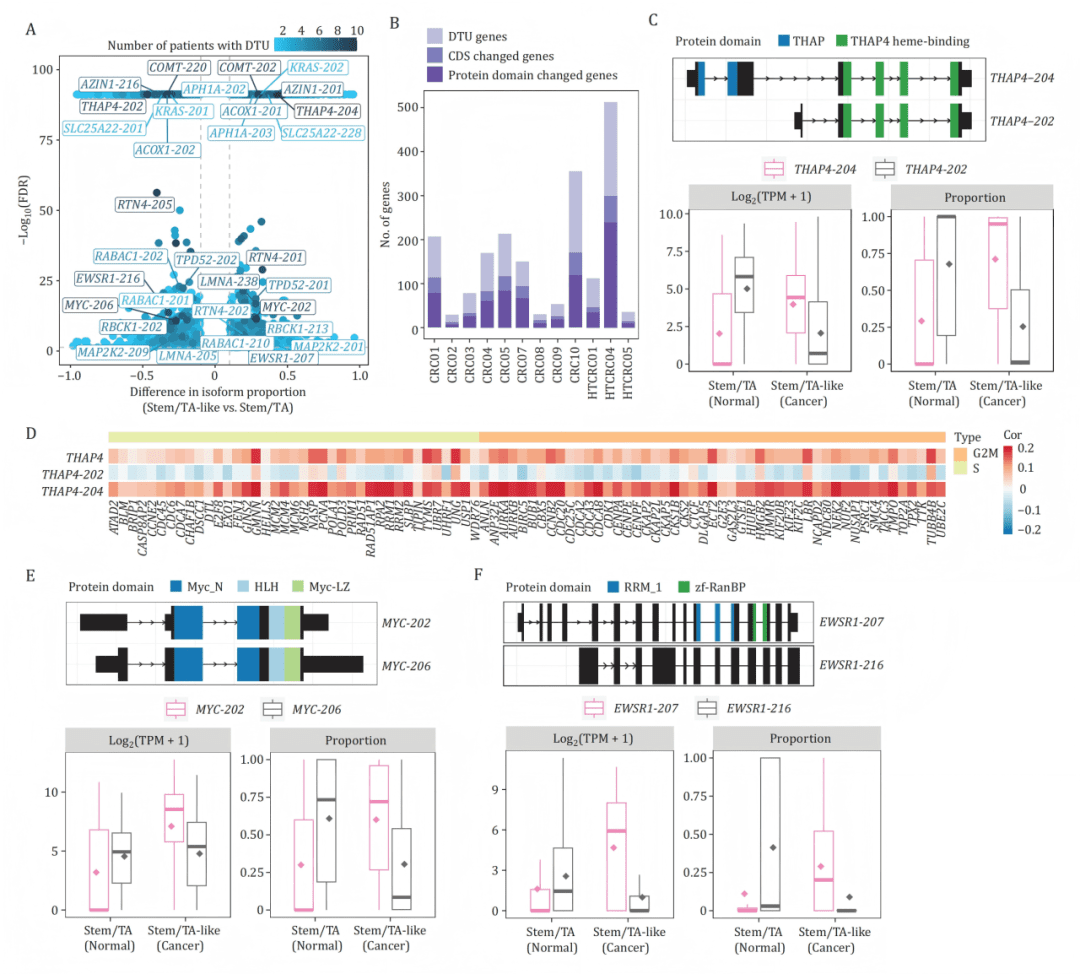
B:多数DTU基因伴随着编码序列及蛋白质结构域的改变。
C-F:以THAP4、MYC、EWSR1为例,详细展示其主导异构体在癌变过程中的转换(C, E, F上)、蛋白质结构域差异(C, E, F下),以及THAP4异构体与细胞周期基因的相关性(D)。
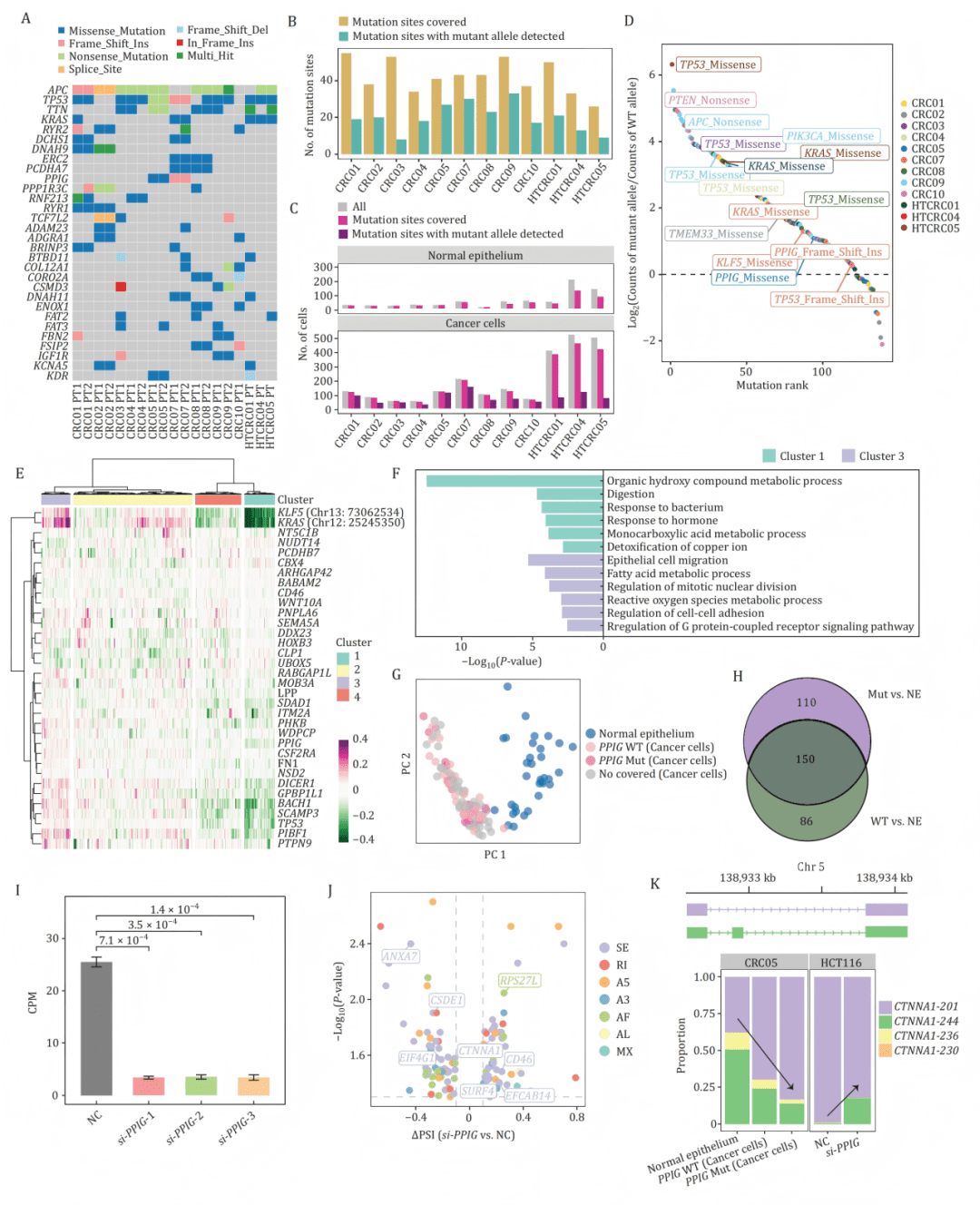
B-C:在单细胞中可检测到突变位点覆盖及突变等位基因表达,且主要存在于癌细胞。
D:点图显示KRAS、TP53等关键基因的突变等位基因在癌细胞中占主导表达。
E-F:在患者CRC07中,KRAS/KLF5突变等位基因的表达与促癌基因簇正相关,而野生型等位基因则与正常功能基因簇相关。
G-J:携带PPIG突变的癌细胞具有独特的剪接图谱;在细胞系中敲低PPIG可逆转部分异常剪接事件(如CTNNA1)。
K:PPIG敲低后,CTNNA1等基因的剪接模式向正常状态回调。
Lu, Ping et al. “Systematic characterization of full-length RNA isoforms in human colorectal cancer at single-cell resolution.” Protein & cell vol. 16,10 (2025): 873-895. doi:10.1093/procel/pwaf049
![]() 若有所获,欢迎点赞、推荐与分享
若有所获,欢迎点赞、推荐与分享
