蛋白质-蛋白质互作检测技术

Co-IP是利用特异性抗体与目标蛋白(诱饵蛋白)结合,然后通过沉淀的方式将整个蛋白复合物从细胞裂解液中分离出来。通过与蛋白A/G偶联的琼脂糖珠或磁珠,可以高效地富集与目标蛋白相互作用的蛋白(猎物蛋白)。
适用场景:
● 验证两个蛋白是否存在相互作用;
● 筛选与目标蛋白互作的候选蛋白。
局限性:
由于沉淀下来的是蛋白质复合物,不能显示蛋白质之间的相互作用是直接还是间接的。可能存在非特异性结合,不适合研究弱或瞬时的相互作用。
酵母双杂交系统基于真核生物转录因子的模块化特性。将目标蛋白与DNA结合结构域(BD)融合(诱饵),待测蛋白与转录激活结构域(AD)融合(猎物)。只有当两个蛋白发生相互作用时,BD和AD才会在空间上接近,从而激活报告基因的表达。
适用场景:
● 大规模蛋白质相互作用组学研究
● 验证蛋白间的直接相互作用
● 筛选药物靶点
蛋白质-DNA互作检测技术
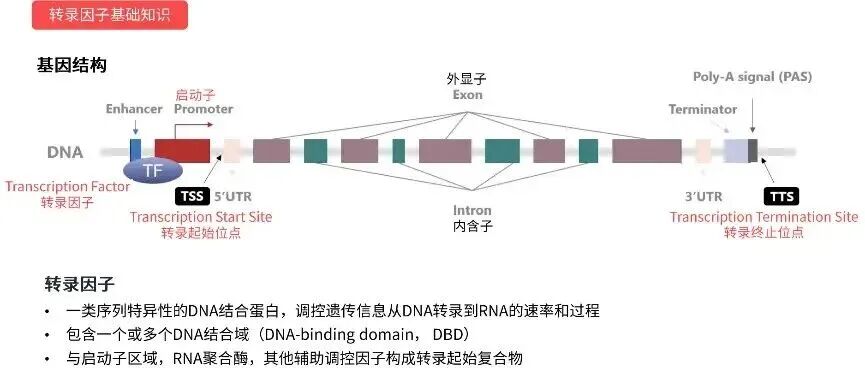
ChIP-seq通过甲醛交联固定蛋白质与DNA的相互作用,使用超声或酶切将染色质片段化,然后用特异性抗体富集目标蛋白结合的DNA片段,最后通过高通量测序确定全基因组范围内的结合位点。
适用场景:
● 转录因子结合位点定位
● 组蛋白修饰(Histone Modifications)图谱绘制
● 有经过验证的高质量、高特异性的抗体
● 拥有相对充足的细胞或组织样本
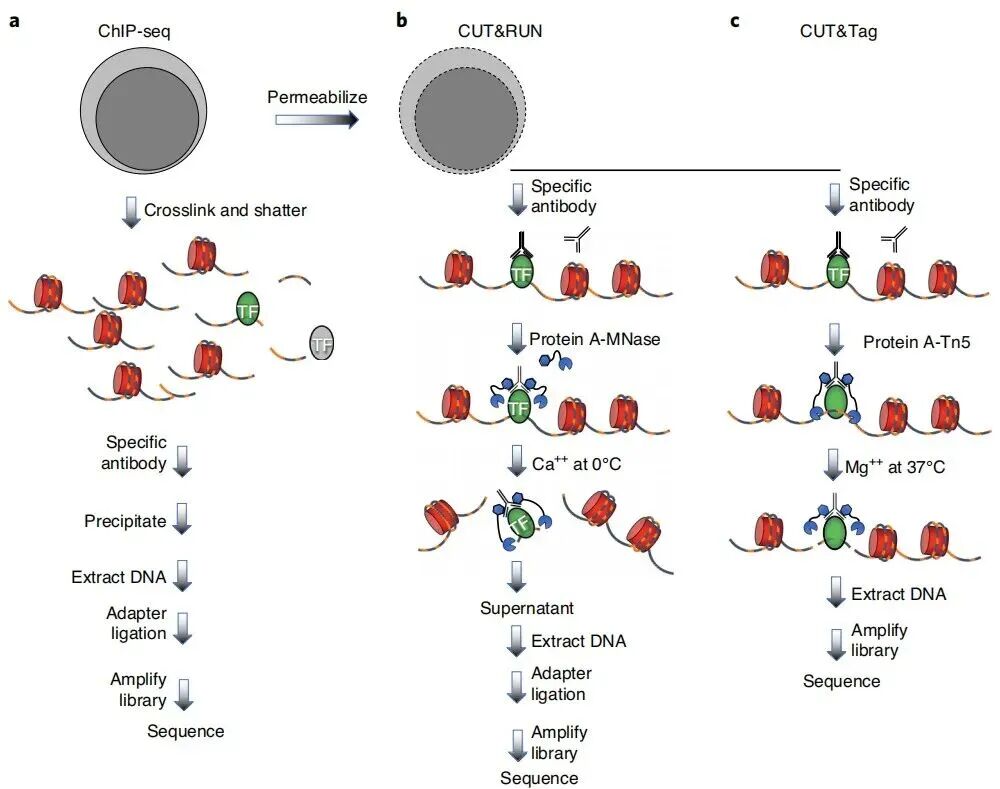
CUT&Tag使用蛋白A-Tn5转座酶融合蛋白在抗体指引下直接对目标蛋白周围的DNA进行切割和标签插入,极大提高了信噪比和实验效率。
适用场景:
● 珍贵或微量样本的研究
● 需要高质量数据和高信噪比的研究
● 快速、高效的高通量筛选
● 研究对交联敏感或难以ChIP的靶标
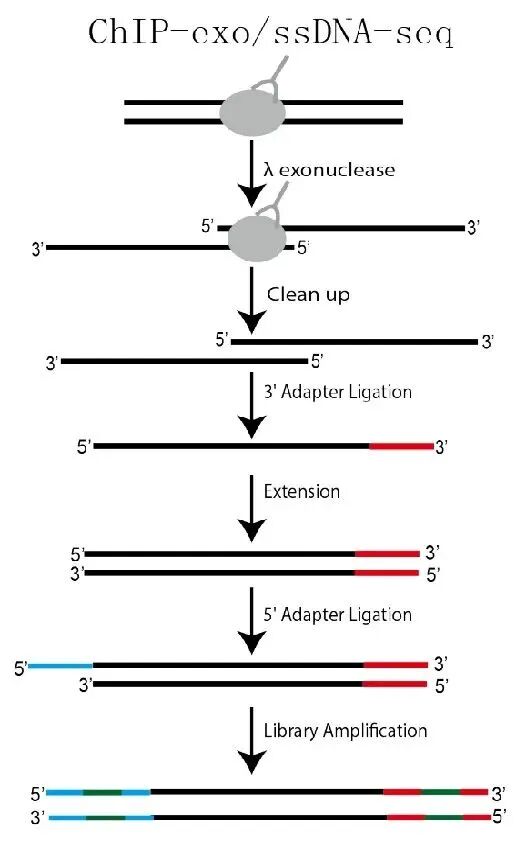
ChIP-exo使用λ-核酸外切酶处理免疫沉淀获得的蛋白质-DNA复合物,λ-核酸外切酶沿5’-3’方向切割DNA双链中的一条链,直到碰到交联蛋白质才停止反应,能够将蛋白质结合位点精确映射到单个核苷酸分辨率。
技术优势:
● 高分辨率:达到单碱基分辨率;
● 特异性强:核酸外切酶消化降低背景DNA;
● 高信噪比:消除检测系统产生的大量噪音;
● 流程简单:优化建库步骤,提高成功率。
适用场景:
● 需要极高分辨率的研究;
● 鉴定转录因子的精确结合序列。
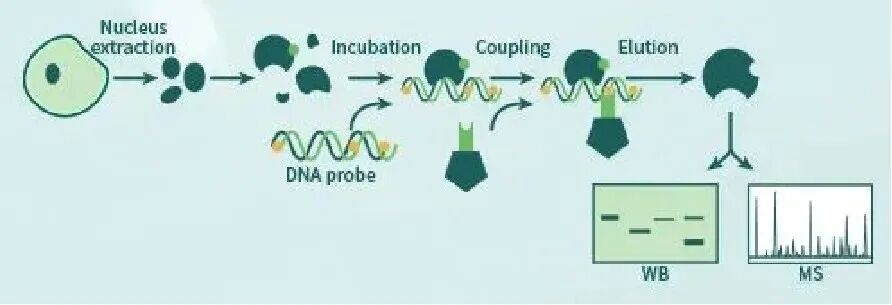
将生物素标记的DNA探针与细胞核提取物孵育,富集结合蛋白后进行鉴定。
适用场景:
● 已知DNA序列(如增强子、启动子),找结合蛋白;
● 验证特定DNA元件与蛋白的互作。
蛋白质-RNA互作检测技术
● RIP-seq:利用抗体捕获蛋白-RNA复合物,测序鉴定结合的RNA;
● iRIP-seq(紫外交联免疫沉淀):通过UV交联固定直接互作,酶切RNA提高分辨率;
● eCLIP-seq:引入Input对照,无需同位素,分辨率更高,更适合直接互作分析。
适用场景:
● 已知蛋白,找其结合的RNA;
● 研究RNA结合蛋白(RBP)的功能。
● 研究经典RNA结合蛋白:iRIP或eCLIP
● 研究非经典RNA结合蛋白:RIP或iRIP
● 研究直接结合的RNA:eCLIP
● 需要高分辨率数据:eCLIP
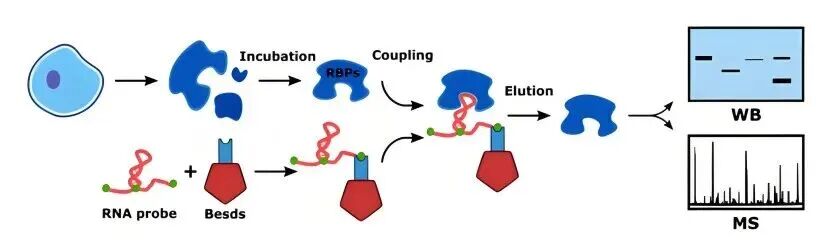
将体外转录的RNA进行生物素标记,与细胞裂解液共同孵育后,通过链霉亲和素磁珠富集与RNA相互作用的蛋白质,最后通过WB或质谱进行鉴定。
适用场景:
● 鉴定与特定RNA结合的蛋白
● 鉴定与特定RNA结合的其他RNA:例如,研究IncRNA与miRNA的相互作用。
● 研究RNA的酬译调控:鉴定与mRNA结合的翻译调控因子。
● 研究RNA的定位:确定与特定RNA结合的定位蛋白。
● 研究RNA在细胞内的定位验证RNA与蛋白质的相互作用。
● 作为其他实验手段(如RIP,CLIP)的体外验证。
通过设计与目标RNA序列反向互补的生物素探针(22 nt),把目标RNA拉下来以后,与其共同作用的DNA染色体片段就会附到链霉亲和素磁珠上,最后通过qRT-PCR或测序来测定目的RNA、DNA序列,亦或是通过Western或质谱分析来测定蛋白。
适用场景:
● 可研究细胞体内互作;
● 既同时可研究RNA、蛋白质和DNA形成的复合体之间的互作;也可研究它们之间的两两互作;
● 寻找与目的lncRNA结合的蛋白,可不受lncRNA长度的限制。
如何选择合适的技术?
● 找RNA →RIP-seq、iRIP-seq、eCLIP-seq(直接结合的用eCLIP-seq)
● 找DNA → ChIP-seq、CUT&Tag、ChIP-exo(样本少选CUT&Tag,要高分辨率选ChIP-exo)
● 找蛋白 →RNA pulldown、ChIRP
● 找DNA → ChIRP
● 找RNA → ChIRP
希望这篇科普能帮助您更好地理解这些技术,并在实际科研中做出更明智的选择。如果您有具体的研究需求,也欢迎进一步交流与合作!
